Miljøprojekt nr. 1135, 2001
Påvisning af patogene bakterier i vand ved anvendelse af DNA-chip-prototype og mikrobiologiske dyrkningsmetoder
Indholdsfortegnelse
1 Formål og valg af modelorganismer
- 2.1 Patogen DNA-chip-prototype: Design og opbygning
- 2.2 Beskrivelse af DNA-chip-analysen
- 2.3 Beskrivelse af de anvendte dyrkningsanalyser
- 2.4 Oversigt over analysetider
- 3.1 Forsøgsplan
- 3.2 Direkte detektion af patogener i suspensionsprøver
- 3.3 Detektion af patogener i spikede vandprøver
4 Prøveforberedelse til DNA-chip-analyse
- 5.1 Fremstilling af suspensionsprøver og prøveforberedelse
- 5.2 Opformering af PCR produkter til DNA-chip-analyse
- 5.3 Resultater opnået med patogen DNA-chip-prototypen
- 5.4 Resultater af prøve A, C og A+C analyseret ved de almindelige dyrkningsmetoder
6 Sammenligning af analysemetoder
Forord
Der er igennem de seneste år udviklet en række forskellige hurtigmetoder til analyse for patogene mikroorganismer i vand. Disse hurtigmetoder er typisk anvendelige til påvisning af en mindre gruppe af bakterier som f.eks. E. coli eller Legionella. Hvis en vandprøve skal screenes for flere patogene bakterier samtidigt, er det nødvendigt at anvende flere forskellige analysemetoder.
Formålet med denne undersøgelse var at analysere, hvorvidt en ny molekylærbiologisk hurtigmetode baseret på DNA-chip-teknologi kan anvendes til hurtig og simultan detektion af mange forskellige vandbårne patogene bakterier, dvs. det er kun nødvendigt at anvende én metode til analyse for flere forskellige bakterier.
Projektet er gennemført som et samarbejdsprojekt mellem Miljøstyrelsens referencelaboratorium Eurofins Danmark A/S og Bioneer A/S. Projektets eksperimentelle arbejde er gennemført i perioden november-december 2005, hvorefter databehandling og diskussion af projektets resultater er foregået inden den endelige afrapportering.
Rapporten beskriver resultatet af undersøgelsen. Rapporten indledes med en kort beskrivelse af projektets formål og valg af modelorganismer. Derefter gennemgås den pågældende DNA-chip-metode og de almindelige mikrobiologiske dyrkningsmetoder, som i projektet er anvendt til påvisning af 5 forskellige vandbårne patogene bakterier. Herefter gennemgås prøveforbehandling og resultater af undersøgelsen, og efterfølgende sammenlignes analyseresultaterne opnået med DNA-chip-metoden og de almindelige dyrkningsmetoder.
Sammenfatning og konklusioner
Baggrunden for denne indledende undersøgelse er, at Bioneer A/S har udviklet en molekylær hurtigmetode baseret på DNA-chip-teknologi til hurtig og simultan detektion af patogene mikroorganismer. ”Proof-of-concept” for anvendelse af en DNA-chip-prototype til påvisning af patogene bakterier er etableret, og næste skridt er at teste anvendeligheden af et sådant koncept i praksis.
Formålet er at undersøge DNA-chip-metodens potentiale og anvendelighed som en hurtig screeningsanalyse for udvalgte patogene mikroorganismer i vand. I projektet sammenlignes DNA-chip-prototypen i dens nuværende form og design (med de begrænsninger prototypen har på specificitet og følsomhed) med almindelige mikrobiologiske dyrkningsmetoder til påvisning og identifikation af patogene bakterier.
Til undersøgelsen er udvalgt 5 modelorganismer. Mikroorganismerne er valgt med den begrundelse, at de kan være årsag til vandbårne sygdomsudbrud samt at de forekommer i vandmiljøer som drikkevand og i badevand. De nævnte mikroorganismer er Campylobacter jejuni, Aeromonas hydrophila, Pseudomonas aeruginosa, Legionella pneumophila og Salmonella spp.. Det eksperimentelle design og forsøgsplanen til undersøgelsen er delt i 2 parallelle blokke, hvor den første del omhandler direkte detektion af patogene bakterier i suspensionsprøver, mens den anden del af forsøget omhandler detektion af patogener spiket i sterilt vand.
DNA-chip-analysen er baseret på en enzymatisk polymerase kæde-reaktion (PCR) opformering af DNA vha. en såkaldt konsensus 16S rDNA-PCR reaktion med endemærkede, ribosomale primere.
Resultatet af DNA-chip-analyse af suspensionsprøver og spikede vandprøver indeholdende blandinger af test-organismer viste, at der opnås de forventede (korrekte) signaler for alle patogene bakterier i prøver bestående af 108 og 106 celler pr. patogen pr. prøve. Konklusionen på de mikrobiologiske analyseresultater er, at dyrkningsmetoderne generelt genfinder de forventede patogene bakterier i både suspensionsprøver og spikede vandprøver indeholdende hhv. 108, 106, 104 og 10² af hver test-bakterie pr. prøve.
Generelt er der overensstemmelse imellem resultaterne opnået med DNA-chip-prototypen og de mikrobiologiske dyrkningsmetoder for bakterieblandingerne indeholdende 108 og 106 celler pr. prøve. DNA-chip-metoden kan i dens nuværende design og assay-format ikke detektere specifikke bakterier ned til 10² celler pr. prøve, men i nogle tilfælde var det muligt at detektere 104 specifikke bakterier. Signaler for positive kontrolprober fås fra alle prøver (også 10²-prøverne) i DNA-chip-analysen.
DNA-chip-metodens følsomhed skal forbedres, hvis metoden skal anvendes til analyse af patogene bakterier i f.eks. drikkevand. Optimering af DNA-chip-metodens følsomhed skal bl.a. gennemføres ved at udvikle en prøve-forbehandlingsmetode, der er målrettet til DNA-analyse af patogene bakterier i vandprøver. I dette projekt var det nødvendigt at anvende en prøveforbehandlingsprocedure, som betød tab af udgangsmateriale, og dermed blev DNA-chip-metodens følsomhed reduceret i forhold til de mikrobiologiske dyrkningsmetoder.
Summary and conclusions
The background for this preliminary investigation is that Bioneer A/S has developed a molecular method based on DNA chip technology for rapid and simultaneous detection of pathogenic microorganisms. This rapid method makes is possible to detect and at the same time identify the various pathogenic bacteria that are relevant in relation to water. Proof-of-concept for use of a DNA chip prototype for detection of pathogenic bacteria is established, and the next step is testing the applicability of the concept.
The aim is to establish the potential and applicability of the DNA chip method as a rapid screening analysis for selected pathogenic microorganisms in water. In the project, the DNA chip prototype in its present form and design (with the prototype's limits concerning specificity and sensitivity) is compared to usual microbiological cultivation methods for detection and identification of pathogenic bacteria.
For this investigation 5 model organisms have been selected because of their capability of causing water-borne diseases and their occurrence in drinking water as well as in bathing water. The organisms are Campylobacter jejuni, Aeromonas hydrophila, Pseudomonas aeruginosa, Legionella pneumophila, and Salmonella spp.
The experimental plan run in two parallel blocks; the first covering direct detection of pathogenic bacteria in suspension samples, the second covering detection of pathogens spiked in sterile water.
The DNA chip analysis is based on an enzymatic polymerase chain reaction (PCR) amplification of DNA by means of a so-called consensus 16S rDNA-PCR reaction with end-labelled ribosomal primers.
The result of DNA chip analysis of suspension samples and spiked water samples containing mixtures of test organisms shows that the expected (correct) signals for all pathogenic bacteria in tests of 108 and 106 cells per pathogen per sample are obtained. Following the microbiological analysis results it is conclusive that the growing methods generally retrieve the expected pathogenic bacteria in suspension samples and spiked water samples containing 108, 106, 104 and 10² cells per sample.
Generally, the results obtained from the DNA chip prototype are in accordance with the results from the microbiological growing methods for the bacterial mixtures containing 108 and 106 cells per sample. In its present design and assay size, the DNA chip method cannot detect specific bacteria as few as 10² cells per sample, but in some cases it was possible to detect 104 specific bacteria.
1 Formål og valg af modelorganismer
1.1 Baggrund og formål
Baggrunden for denne indledende undersøgelse er, at Bioneer A/S har udviklet en molekylær hurtigmetode baseret på DNA-chip-teknologi til simultan detektion af forskellige patogene mikroorganismer. Denne hurtigmetode gør det i princippet muligt at påvise og samtidig identificere de patogene bakterier, der er relevante i relation til vand. ”Proof-of-concept” for anvendelse af en DNA-chip-prototype til påvisning af patogene bakterier er etableret, og næste skridt er at teste anvendeligheden af et sådant koncept i praksis.
Formålet med dette forprojekt er at undersøge DNA-chip-metodens potentiale og anvendelighed som en hurtig screeningsanalyse for 5 udvalgte patogene mikroorganismer i vandprøver.
Det skal bemærkes, at der til dette forprojekt er anvendt en DNA-chip-prototype, som ikke er færdigudviklet. Den nuværende udgave af DNA-chip-prototypen har begrænsninger mht. specificitet samt indeholder nogle ekstra DNA-kontroller, der har vist sig ikke at fungere optimalt i test-forsøg. Disse begrænsninger har ingen betydning for konklusionerne i dette projekt, og DNA-kontrollerne vil blive korrigeret på næste version af DNA-chippen.
1.2 Valg af mikroorganismer
Mikroorganismerne er valgt med den begrundelse, at de kan være årsag til vandbårne sygdomsudbrud samt at de forekommer i vandmiljøer som drikkevand og i badevand. De nævnte mikroorganismer er Campylobacter jejuni, Aeromonas hydrophila, Pseudomonas aeruginosa, Legionella pneumophila og Salmonella spp.. Mikroorganismerne er opdelt i følgende tre grupper:
- Patogene bakterier relevante for drikkevand (Drikkevandsgruppen): Campylobacter jejuni, Aeromonas hydrophila og Pseudomonas aeruginosa.
- Patogene bakterier relevante for varmtvand (Varmtvandsgruppen): Legionella spp.
- Patogene mikroorganismer relevante for diverse vandmiljøer (Diverse-gruppen): Campylobacter jejuni/coli, Salmonella spp. og Legionella pneumophila, serotype 1.
Legionella er eneste mikroorganisme i gruppe B, og det blev besluttet ikke at analysere gruppe B særskilt, fordi slægten Legionella også indgår i gruppe C.
Til de eksperimentelle analyser i denne undersøgelse er der derfor valgt følgende tre blandinger af bakteriekulturer:
| Blanding A): | "Drikkevandsgruppen; Campylobacter jejuni + Aeromonas hydrophila + Pseudomonas aeruginosa. |
| Blanding C): | Diverse-gruppen; Campylobacter jejuni + Salmonella spp. + Legionella pneumophila, serotype 1. |
| Blanding A+C): | Alle. |
Det eksperimentelle design i undersøgelsen er beskrevet i afsnit 3.
2 Analysemetoder
- 2.1 Patogen DNA-chip-prototype: Design og opbygning
- 2.2 Beskrivelse af DNA-chip-analysen
- 2.3 Beskrivelse af de anvendte dyrkningsanalyser
- 2.4 Oversigt over analysetider
Denne indledende undersøgelse har fokus på at sammenligne en ny DNA-chip-metode (prototype) med de gældende mikrobiologiske dyrkningsmetoder. I dette afsnit gives en detaljeret beskrivelse af patogen DNA-chip-metoden samt en kort beskrivelse af analyseprincip og analysetider for de mikrobiologiske dyrkningsmetoder, som er anvendt i projektarbejdet.
2.1 Patogen DNA-chip-prototype: Design og opbygning
Bioneer A/S har udviklet en DNA-chip-prototype til simultan detektion af forskellige patogene mikroorganismer. Patogen-DNA-chippen består af et glas-slide, hvorpå der er påsat i alt 24 DNA-prober, som har en længde på mellem 17-23 nukleotider. DNA-proberne er designet udfra bakteriers 16S ribosomale DNA (rDNA), hvilket gør det muligt at opbygge et hierarkisk design af DNA-prober på chippen, som sikrer, at der for hver enkelt bakterie-art detekteres positivt signal fra flere forskellige prober, dvs. en slags intern kvalitetskontrol.
Oversigt over udvalgte bakterier med tilhørende DNA-prober er givet i tabel 1.
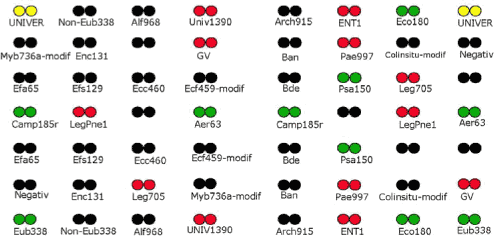
Tabel 1: Liste over bakterier med tilhørende 16S rDNA-probe, som er anvendt til udvikling af DNA-chippen (prototype). I kolonnen med 16S rDNA prober er markeret, hvilke prober der aktuelt er anvendt til detektion af de 5 udvalgte patogener i projektet. Hver enkelt DNA-probe på DNA-chippen giver enten Cy3 (grønne signaler) eller Cy5 (røde signaler) alt afhængig af, hvilket af de 2 PCR produkter, som DNA-proben hybridiserer til (se figur 2). De forventede signal-farver for de enkelte DNA-prober er angivet i tabellen med et kryds.

Som det fremgår af tabel 1 veksler probernes specificitetsniveau fra at være specifikke for få arter (f.eks. Psa150 og Camp185r) til at være slægtsspecifikke (f.eks. Aer63 og Leg705) eller generelle (f.eks. UNIVER, Univ1390 og Eub338). Specificitetsniveauet er bestemt ud fra sekvens-analyser med anvendelse af internationale databaser, som indeholder 16S ribosomale DNA-sekvenser fra tusindvis af bakterier.
I projektet anvendtes Camp185r-DNA-proben til påvisning og identifikation af Campylobacter jejuni, men denne DNA-probe kan også detektere C. coli og C. lari, jf tabel 1. Der er 2 prober på DNA-chip-prototypen til detektion af Pseudomonas aeruginosa (Psa150 og Pae997), og ligeledes 2 DNA-prober til detektion af Legionella pneumophila, serotype 1 (LegPne1 og Leg705). Aeromonas hydrophila påvises ved hjælp af Aeromonas-slægtsproben (Aer63). DNA-proben for Enterobacteriaceae (Eco180) anvendes til detektion af Salmonella i dette projekt, fordi DNA-chip-prototypen ikke indeholder en Salmonella-specifik probe.
Tidligere test-forsøg har vist, at Enterobacteriaceae-proben ENT1 ikke virker, hvorfor denne probe ikke inddrages i projektet.
De 3 DNA-prober UNIVER, Univ1390 og Eub338 er anvendt som positive kontroller på DNA-chippen.
I figur 1 er placeringen af de enkelte 16S rDNA-prober på chippen angivet. Patogen DNA-chippen har en standard opbygning med positive kontrol-prober i alle 4 hjørner, der giver positive signaler for hhv. alle bakterier (UNIVER) og Eu-bakterier (Eub338). Derudover er kontrolproben Univ1390 placeret midt på øverste og nederste række, jf. figur 1.
Hver DNA-probe er spottet på DNA-chip-prototypen i duplikat i 2 forskellige positioner, dvs. hver DNA-probe er repræsenteret med 4 spots pr. chip.
Figur 1: Placering af 16S rDNA-prober på patogen DNA-chip (prototype). DNA-prober til detektion af de 5 udvalgte patogene bakterier i projektet samt kontrol-prober er markeret med gul/grøn/rød farve svarende til det forventede signal på DNA-chippen (se tabel 1 og figur 2). Alle øvrige prober er markeret med sort. Spots uden probe-navn er tomme positioner på DNA-chippen.
Fremstillingen af patogen DNA-chips foregik i praksis ved, at DNA-proberne blev spottet på Epoxid-coatede glasslides (Corning) ved hjælp af en Genetix Q-Pix microarrayer.
2.2 Beskrivelse af DNA-chip-analysen
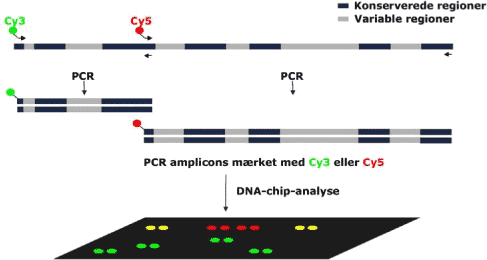
Udgangspunktet for selve DNA-chip-analysen er en enzymatisk polymerase kæde-reaktion (PCR) opformering af DNA vha. en såkaldt konsensus 16S rDNA-PCR reaktion med endemærkede, ribosomale primere.
I praksis gennemføres PCR reaktionen ved amplifikation af hhv. et 530 bp fragment (mærket med Cy3) og et 1000 bp fragment (mærket med Cy5), der tilsammen dækker hele 16S rDNA-regionen (illustreret som den øverste stang i figur 2). De 2 fluorescens-mærkede PCR produkter oprenses, blandes og anvendes til hybridisering til DNA-chippen (figur 2). Resultatet af analysen visualiseres ved fluorescens-scanning, f.eks. på en ArrayWoRx scanner (Applied Precision).
Hvis en given bakterie er til stede i prøven, vil de tilsvarende DNA-prober immobiliseret på DNA-chippen give et positivt Cy3- eller Cy5-signal afhængig af den pågældende DNA-probes placering, dvs. mærkning med to forskellige signal-molekyler giver en intern grøn/rød kvalitetskontrol for diskrimination mellem korrekte og falske-positive signaler i assayet. Gule spots opstår som et mix af grøn og rød, fordi de to PCR produkter hybridiserer lige effektivt til den samme DNA-probe. Dette er tilfældet for en enkelt probe (UNIVER) i det nuværende design af DNA-chippen, fordi proben har et overlap med begge PCR produkter.
Figur 2: Skematisk fremstilling af detektionsprincippet i DNA-chip-analysen.
Patogen-DNA-chippen har været testet under en lang række kontrollerede betingelser, hvor DNA-chippens evne til at give et korrekt respons på blandingsprøver af renkulturer bestående af 2-7 forskellige bakteriearter har været undersøgt og valideret inden starten på dette projekt.
Anvendelse af DNA-chip-metoden til påvisning af i alt 5 patogene bakterier i blanding A, C og A+C (jf afsnit 1.2) er beskrevet i afsnit 3.
2.3 Beskrivelse af de anvendte dyrkningsanalyser
Analyseprincip, arbejdsgangen for at nå frem til det færdige resultat og analysetid er kort beskrevet for hver enkelt mikrobiologisk dyrkningsmetode således, at det er muligt at foretage en samlet vurdering af fordele og ulemper ved DNA-chip-analysen sammenlignet med de mikrobiologiske metoder på baggrund af de metode-tekniske oplysninger i dette afsnit.
2.3.1 Bestemmelse af mesofile Aeromonas arter: NMKL 150:2004
Analyseprincip: Kvantitativ bestemmelse af Aeromonas foretages ved overfladeudsæd af prøven på Stivelse-Ampicillin Agar (SAA) og inkubering ved 37,0°C i 24 timer.
Aflæsning: Aeromonas suspekte kolonier ses som gule eller honningfarvede kolonier med en diameter på 2-3 mm. Kolonierne er omgivet af en lys zone med en bredde på 2-3 mm. Verifikation foretages efter rendyrkning på Luria Bouillon agar(LB agar) uden og med 6% salt. LB-agarpladerne inkuberes ved 37,0°C i 24 timer både aerobt og anaerobt. Før inkubation tilsættes den aerobe skål vibriostatica.
Analysetid: Bestemmelse af Aeromonas-arter efter NMKL 150:2004 tager 2 døgn, inklusiv verifikation af kolonierne, før det endelig resultat foreligger.
2.3.2 Bestemmelse af Pseudomonas aeruginosa: DS 268:1990
Analyseprincip: Kvantitativ bestemmelse af Pseudomonas aeruginosa foretages ved membranfiltrering af prøven, hvorefter filteret anbringes på overfladen af selektivt agar Cetrimid-nalidixinsyre-agar (CN-agar) og inkuberes ved 42°C i 48 timer.
Aflæsning: Fremvoksede kolonier tælles. Pladen anbringes under UV-lys, og der foretages verifikation af fluoriserende kolonier ved subkultivering på mælkeagar. Pladerne inkuberes ved 42°C i 24 timer. Pseudomonas aeruginosa er katalase- og oxidase-positive, nedbryder glucose oxidativt, hydrolyserer kasein (klar zone omkring kolonien på mælkeagar) og danner fluoriserende kolonier.
Analysetid for påvisning og aflæsning af Pseudomonas aeruginosa er 2 døgn plus 1 døgn til subkultivering/verifikation, dvs. i alt 3 døgn, før det endelig resultat foreligger.
2.3.3 Bestemmelse af termofile Campylobacter: ISO 17995:2005
Analyseprincip: Kvalitativ bestemmelse af termofile Campylobacter foretages ved, at prøven membranfiltreres. Filtrene overføres til to selektive opformeringsbouilloner (Bolton bouillon og Preston bouillon), der inkuberes ved 37°C i 2 døgn i mikroaerofil atmosfære. Efter inkubation udstryges fra hver bouillon på overfladen af et selektivt fast substrat, modificeret Charcoal Cephoperazone Desoxycholate Agar, mCCDA, der inkuberes ved 41,5°C i 2 døgn i mikroaerofil atmosfære.
Aflæsning: Suspekte kolonier undersøges for vækst under aerobe forhold. Isolater, der ikke vokser aerobt, undersøges ved mikroskopi. Herefter kan enkeltliggende kolonier verificeres ved subkultivering på blodagarplader med efterfølgende inkubering aerobt og anaerobt ved 41,5°C i 1 døgn. Kolonierne undersøges ved mikroskopi. Campylobacter vokser ikke aerobt, men mikroaerofilt. Campylobacter er stærkt bevægelige, spiralformede (proptrækker-agtige) stave.
Analysetid for termofile Campylobacter inkluderer et selektivt præopformeringstrin på 2 døgn. Den samlede analysetid for termofile Campylobacter er 4 døgn plus ét døgn til verifikation, dvs. i alt 5 døgn, før det endelig resultat foreligger.
2.3.4 Bestemmelse af Salmonella: DS 266:1988
Analyseprincip: Kvalitativ bestemmelse af Salmonella foretages ved, at prøven membranfiltreres. Filtrene overføres til non-selektiv opformeringsbouillon (buffered pepton vand, BPW), der inkuberes ved 37°C i 16-24 timer. Herefter overføres blandingen til 2 selektive opformeringsbouilloner (Rappaport Vassiliadis (RV) bouillon og Müller-Kauffmann bouillon (MK)), der inkuberes ved 42°C i 24 timer. Efter inkubation udstryges fra opformeringen på overfladen af selektive plader, brilliantgrønt-laktose-saccharose-phenolrødt-agar (BLSF) og phenolrødt-mannitol-agar. Pladerne inkuberes ved 37°C i 24 timer.
Aflæsning: På BLSF-agar er typiske kolonier transparente til rødlige, substratet skifter farve fra lys orange til rød. På phenolrødt-mannitol-agar er typiske kolonier gule. Suspekte kolonier rendyrkes på PCA (Plate Count Agar), hvorefter der foretages biokemisk og serologisk verifikation af disse kolonier.
Analysetid: Metoden til påvisning af Salmonella inkluderer to præ-opformeringstrin samt rendyrkning af suspekte kolonier. Den samlede analysetid er 5 døgn, inkl. verifikation, før det endelig resultat foreligger.
2.3.5 Bestemmelse af Legionella: DS 3029:2001
Analyseprincip: En aliquot af prøven (0,1 ml) udsås direkte på GVPC agar (Modificeret Charcoal Yeast Extract agar med selektivt supplement). Den resterende prøve opkoncentreres dels ved membranfiltrering (100x opkoncentrering), dels ved efterfølgende centrifugering (i alt 1000x opkoncentrering) og udsås derefter på GVPC. Pladerne inkuberes ved 37°C. Såfremt pladerne ikke kan aflæses pga. baggrundsvækst udføres syre- og varmebehandling af prøverne før fornyet udsæd.
Aflæsning: GVPC-pladerne aflæses efter ca. 4 og ca. 7 dage. Slutaflæsning foretages efter 10 dage. De fremkomne kolonier tælles og verificeres. Typiske kolonier er konvekse, helrandede, glinsende med kornet overflade. Grå, hvide, blå-violette eller lime-grønne. Nogle kan fluorescere. Kolonierne er små (pin-point) efter 2 – 3 dage, men vokser derefter til ca. 3 – 4 mm. Suspekte kolonier rendyrkes på PCA, hvorefter der foretages biokemisk og serologisk verifikation.
Analysetid for bestemmelse af Legionella er 10 døgn plus 2 døgn til verifikation, før det endelig resultat foreligger.
2.4 Oversigt over analysetider
Overordnet set er der meget stor forskel på den samlede analyse- og verifikationstid for de mikrobiologiske dyrkningsmetoder, jf tabel 2.
Tabel 2: Oversigt over analysetid (døgn) og verifikationstid (døgn) for de anvendte mikrobiologiske dyrkningsmetoder.
| Analysetid, døgn | Verifikation, døgn | |
| Aeromonas spp. | 1 | 1 |
| Pseudomonas aeruginosa | 2 | 1 |
| Campylobacter spp. | 4 | 1 |
| Salmonella spp. | 3 | 2 |
| Legionella spp. | 10 | 2 |
Til sammenligning kan DNA-chip-metoden anvendes til analyse og verifikation af alle 5 udvalgte patogene bakterier inden for 1 døgn.
3 Eksperimentelt design
- 3.1 Forsøgsplan
- 3.2 Direkte detektion af patogener i suspensionsprøver
- 3.3 Detektion af patogener i spikede vandprøver
3.1 Forsøgsplan
Det eksperimentelle design til undersøgelsen er delt i 2 parallelle blokke, hvor den første del omhandler direkte detektion af patogene bakterier i suspensionsprøver (dvs. uden prøveforberedelse), mens den anden del af forsøget omhandler detektion af patogener spiket i sterilt vand. Prøveforberedelse i form af et membranfiltrerings-trin er inkluderet i analysen af vandprøverne, som skitseret nedenfor.
FORSØGSPLAN
| Fremstilling af suspensionsprøver af blanding A, C og A+C Analyse af prøver vha. DNA-chip og mikrobiologiske referencemetoder (ingen prøveforberedelse) |
Fremstilling af spikede vandprøver tilsat blanding A, C og A+C Analyse af prøver vha. DNA-chip og mikrobiologiske referencemetoder (med prøveforberedelse) |
3.2 Direkte detektion af patogener i suspensionsprøver
Først blev hver af de 5 patogene bakterie-isolater podet i et optimalt vækstmedium med henblik på fremstilling af overnatskulturer. Dernæst blev koncentrationen (bakterieceller/ml) af de enkelte overnatskulturer bestemt ved pladespredning/tælling i tællekammer, og suspensionskultur-blandinger af A, C og A+C indeholdende hhv. 108, 106, 104 og 10² bakterieceller af hver af testorganismerne blev fremstillet efter nedenstående skema. Eksempelvis blev der tilsat i alt 106 bakterieceller af Campylobacter jejuni, 106 bakterieceller af Aeromonas hydrophila og 106 bakterieceller af Pseudomonas aeruginosa til suspensionsprøve A (106). Indholdet af bakterier i blandingerne A, C og A+C er angivet nedenfor og i afsnit 1.2.
| Blanding A): | Campylobacter jejuni + Aeromonas hydrophila + Pseudomonas aeruginosa. |
| Blanding C): | Campylobacter jejuni + Salmonella spp. + Legionella pneumophila, serotype 1. |
| Blanding A+C): | Alle. |
Suspensionsprøverne blev efterfølgende analyseret efter de mikrobiologiske dyrkningsmetoder beskrevet i afsnit 2. Parallelt hermed blev suspensionsprøverne også analyseret ved anvendelse af DNA-chip-prototypen. De herved opnåede samlede analyseresultater er gennemgået i afsnit 5.
Tabel 3: Oversigt over antallet af suspensionsprøver fremstillet til hhv. mikrobiologisk analyse og DNA-chip-analyse. De mikrobiologiske dyrkningsmetoder kræver flere analyser i de lave koncentrationer, hvorfor der er forskel på antallet af prøver.
| Antal celler pr. patogen |
Antal suspensionsprøver til mikrobiologisk analyse |
Antal suspensionsprøver til DNA-chip-analyse |
| 108 | 2 A, 2 C, 2 A+C | 2 A, 2 C, 2 A+C |
| 106 | 2 A, 2 C, 2 A+C | 2 A, 2 C, 2 A+C |
| 104 | 8 A, 8 C, 8 A+C | 2 A, 2 C, 2 A+C |
| 10² | 10 A, 10 C, 10 A+C | 2 A, 2 C, 2 A+C |
3.3 Detektion af patogener i spikede vandprøver
Spikede vandprøver blev fremstillet ved, at sterilt vand (100 ml) blev spiket med blanding A, C og A+C indeholdende hhv. 108, 106, 104 og 10² bakterieceller for hver af de udvalgte patogene testorganismer (jf. 1.2 og 3.1).
I praksis foregik det ved, at der blev fremstillet endnu 4 ekstra sæt af bakterieblandingerne A, C, og A+C for hver koncentration. Herefter blev bakterieblandingerne fortyndet i sterilt vand til et slutvolumen på 100 ml, som blev filtreret igennem et 0.45 µm filter. Dette prøveforbehandlingstrin resulterede i, at bakterierne blev opsamlet på et filtermateriale, som herefter blev anvendt til analyse med hhv. mikrobiologiske dyrkningsmetoder og DNA-chip-metoden.
3.3.1 Valg af filtermateriale
Til de mikrobiologiske referenceanalyser anvendtes et standard Millipore MF filter, type HAWG (=mixed cellulose acetate og cellulose nitrat, porestørrelse 0.45 µm, filterdiameter 47 mm).
HAWG filtermateriale kan ikke anvendes til DNA-forsøg, fordi denne filtertype har en relativ høj uspecifik bindingskapacitet, idet DNA, protein, cellerester og lignende bindes til filteret. Desuden anvendes detergent (Triton) til fremstilling af filteret, som inhiberer DNA-chip-analysen.
Spikede vandprøver, som skulle analyseres ved hjælp af DNA-chip-prototypen, blev derfor i stedet filtreret igennem et Durapore HVLP filtermateriale (Millipore) med samme porestørrelse og diameter som MF-filtermaterialet til de mikrobiologiske referencemetoder. Durapore-materialet minder i opbygning og egenskaber om et teflon-filter, (dog med 2 i stedet for 4 Fluor-radikaler påsat), og der er ikke brugt detergent under fremstillingsproceduren. Tidligere undersøgelser har vist, at Durapore ikke har nogen signifikant inhiberende effekt på PCR, dvs. det er muligt at anvende Durapore-filtermaterialet direkte i en DNA-analyse (Oyofo og Rollins, 1993. Appl Environ Microbiol. 59(12):4090-5).
4 Prøveforberedelse til DNA-chip-analyse
Første trin i prøveforbehandling er ekstraktion af DNA i en form, som gør det muligt at gennemføre en PCR- og DNA-chip-analyse. Til lysering af bakterier anvendes tilsætning af 0.006N NaOH og varmebehandling ved 95°C. Den lyserede prøve kan anvendes direkte til PCR, dvs. der undgås et tidskrævende DNA-oprensningstrin, hvor der kan mistes udgangsmateriale undervejs. I denne undersøgelse er den ovenfor nævnte lyseringsmetode anvendt til lysis af cellerne i alle prøver, og prøverne er herefter direkte anvendt til PCR opformering af ribosomalt DNA efterfulgt af DNA-chip-analyse, jf. figur 3.
Figur 3: Flow-diagram over prøveforbehandling til DNA-chip-analyse af hhv. suspensionsprøver og filtermateriale fra spikede vandprøver.
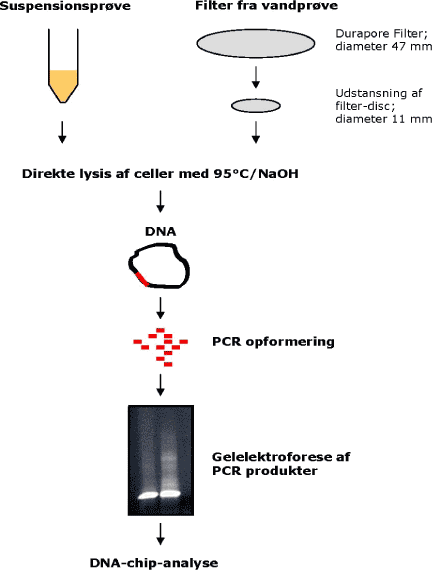
Som det fremgår af figur 3 verificeres PCR-produkterne ved agarosegel-elektroforese før DNA-chip-analyse.
Til analyse af ”vandprøverne” (sterilt vand spiket med blanding A, C og A+C) blev der indledningsvist testet 4 forskellige prøveforbehandlingsmetoder, idet DNA-chip-analysen ikke tidligere har været testet med et filter som prøvemateriale, hvor filterdiameteren var større end 1 cm. Resultaterne af disse indledende tests viste, at den mest effektive prøveforbehandlingsteknik var direkte lysis af cellerne på filteret med 95°C/0.006N NaOH, hvilket i praksis blev testet ved udstansning af en mindre filterdisc, som herefter kunne anvendes til lysis og PCR (figur 3).
Ulempen ved denne fremgangsmåde er, at der kun analyseres på et mindre areal af filteret: Filter-disc (11 mm) udgør 7,6% af totalt filterareal (diameter estimeret til 40 mm, når filterkanten uden påsat prøve er klippet fra), hvilket giver et tab i analysens følsomhed.
5 Resultater
- 5.1 Fremstilling af suspensionsprøver og prøveforberedelse
- 5.2 Opformering af PCR produkter til DNA-chip-analyse
- 5.3 Resultater opnået med patogen DNA-chip-prototypen
- 5.4 Resultater af prøve A, C og A+C analyseret ved de almindelige dyrkningsmetoder
5.1 Fremstilling af suspensionsprøver og prøveforberedelse
Suspensionskultur-blandinger af A, C og A+C indeholdende hhv. 108, 106, 104 og 10² celler af hver af testorganismerne blev fremstillet, som beskrevet i forsøgsplanen (afsnit 3), bortset fra enkelte afvigelser, som er beskrevet i dette afsnit.
Udgangsmaterialet for forsøget er overnatskulturer af hver af de 5 testorganismer. Ved forsøgsstart viste det sig, at start-kulturen af Campylobacter jejuni, Aeromonas hydrophila og Legionella pneumophila ikke var vokset op til den forventede celletæthed, hvilket gav mangel på materiale i forsøget. Som resultat heraf måtte nogle prøver udgå; således indeholder prøve A (108) kun 108 celler af Pseudomonas aeruginosa, og ikke 108 celler af hhv. Campylobacter jejuni og Aeromonas hydrophila. På tilsvarende vis blev der ikke tilsat Campylobacter jejuni til prøve C (108) og til halvdelen af prøverne A+C (108). Dette gælder for både suspensionsprøver og spikede vandprøver.
Endvidere betød mangel på materiale, at det ikke var muligt at gennemføre DNA-chip-analyse direkte på suspensionsprøver uden prøveforberedelse, som der ellers var lagt op til i den oprindelige forsøgsplan. Suspensionsprøverne skulle efter planen have haft et max. volumen på ca. 60 µl, men dette viste sig ikke at være praktisk muligt, fordi celletætheden af udgangsmaterialet var for lav. Det blev derfor nødvendigt at inkludere et ekstra opkoncentreringstrin (centrifugering) af suspensionsprøverne inden DNA-analyse for at reducere prøvevolumen fra ca. 250 µl til 60 µl. Dette ekstra opkoncentreringstrin vil betyde et tab af materiale (primært for Campylobacter, som er vanskelige at spinde ned), og dermed vil DNA-chip-metodens følsomhed blive reduceret.
Til prøveforbehandling af filtermateriale til DNA-chip-analyse blev der i projektet valgt en procedure med direkte anvendelse af filtermaterialet i PCR. Denne prøveforbehandling kan anvendes på næsten alle bakterier, og prøveforbehandlingen tager kun 10 minutter.
Ulempen ved denne metode er, at prøveforbehandlingen til DNA-chip-analysen på nuværende tidspunkt kun er skaleret til filterdiscs med en diameter på max. 11 mm. Det betyder, at der kun anvendes 7,6 % af det totale filterareal til DNA-chip-analysen, hvilket giver mere end en 10x faktors forringelse af analysens følsomhed. Prøveforbehandlingsteknikken kan opskaleres til at kunne håndtere større filtermaterialer eller der kan anvendes alternative forbehandlingstrin, men et sådant udviklingsarbejde lå ikke inden for rammerne af dette projekt.
5.2 Opformering af PCR produkter til DNA-chip-analyse
Som det fremgår af figur 2 (se afsnit 2.2) foregår en DNA-chip-analyse ved, at ét kort PCR produkt (530 bp) og ét langt PCR produkt (1000 bp) blandes og hybridiseres til DNA-chippen, dvs. der skal fremstilles to PCR produkter fra hver prøve til DNA-chip-analysen.
Alle PCR-produkter visualiseres og kontrolleres ved agarosegelelektroforese inden brug (figur 3). Resultatet af en agarosegelelektroforese illustreret i figur 4 afspejler det generelle resultat for alle PCR-opformeringer af suspensions- og filterdisc-prøver for 530 bp-fragmentet i projektet: Specifikke PCR-produkter af korrekt størrelse blev amplificeret fra A, C og A+C-prøver indeholdende 108 og 106 celler, mens det ikke var muligt at detektere PCR-produkterne fra 104 og 10² celler ved agarosegelelektroforese, dvs. PCR produkterne var til stede i en koncentration, der ligger under detektionsgrænsen på en agarosegel.
Figur 4: Billede af PCR produkter (530 bp fragmenter) efter agarosegel-elektroforese, hvor produkterne kontrolleres inden videre brug til DNA-chip-analyse. (Prøven ”M” længst til højre på gelen er en størrelsesmarkør).
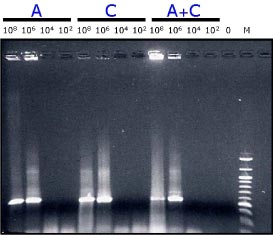
Dette resultat er i overensstemmelse med, at detektionsgrænsen for 16S konsensus PCR-assayet på rent DNA er ca. 10³-104 celler i en agarosegel (se bilag A) samt at prøveforbehandlingen, (dvs. en ekstra centrifugering af suspensionsprøver og anvendelse af mindre filter-disc fra spikede vandprøver) medfører en reduktion af metodens følsomhed.
5.3 Resultater opnået med patogen DNA-chip-prototypen
5.3.1 Kontrolforsøg
Før gennemførelse af DNA-chip-analyse af suspensionsprøver og vandprøver blev DNA-chippen testet i kontrolforsøg: Der blev oprenset rent DNA fra de 5 patogene bakterier i undersøgelsen og test af PCR/DNA-chip-analyse blev udført på kontrolprøver indeholdende kun én patogen samt på prøver indeholdende udvalgte blandinger af de 5 patogene bakterier. Resultaterne viste, at der generelt opnås de forventede signaler med patogen DNA-chip-prototypen.
I enkelte tilfælde kan der forekomme krydshybridisering mellem PCR-produkt fra Legionella og DNA-probe GV for slægten Vibrio. Desuden gav Legionella-DNA-proben generelt svagere signaler på DNA-chip-prototypen end de øvrige prober. Der skal derfor optimeres på Legionella/Vibrio-detektionen før prototypen kan anvendes til specifik detektion af begge disse 2 patogener.
5.3.2 Analyse af prøve A, C og A+C
Figur 5 og 6 viser nogle eksempler på de resultater, der er opnået med patogen DNA-chip-prototypen til analyse af bakterieblanding A, C og A+C.
Som allerede nævnt har patogen DNA-chip-prototypen en standard opsætning med positive kontrol-prober i alle 4 hjørner, der giver positive signaler for hhv. alle bakterier (2 gule spots fra UNIVER-proben i de øverste hjørner) og Eu-bakterier (2 grønne spots fra Eub338-proben i de nederste hjørner), jf. figur 5. De øvrige røde spots på øverste og nederste række på DNA-chippen i figur 5 er signaler fra Univ1390-kontrolproben (til venstre) og et falsk positivt signal fra proben for Archae-bakterier (til højre), som er velkendt fra tidligere testforsøg med denne prototype.
Resultatet af DNA-chip-analyse af suspensionsprøve A+C (108) er illustreret i figur 5, hvor den øverste del af figuren viser et billede af selve DNA-chippen, mens den tilhørende databehandling er illustreret nederst. Af DNA-chippen øverst ses, at der opnås de forventede (korrekte) signaler for alle patogene bakterier i denne prøve A+C (108) (fra venstre på billedet): Legionella pneumophila /Legionella sp. (blå kasser), Aeromonas spp. (blå cirkler), Pseudomonas aeruginosa (hvide cirkler) og Salmonella spp. (hvide kasser).
Figur 5: Resultat af DNA-chip-analyse, hvor patogen DNA-chip-prototypen er anvendt til analyse af suspensionsprøve A+C (108). Prøven indeholder følgende bakterier: Aeromonas spp., Pseudomonas aeruginosa, Salmonella spp. og Legionella pneumophila, serotype 1. Campylobacter jejuni blev ikke tilsat pga. materiale-mangel, jf. afsnit 5.1.
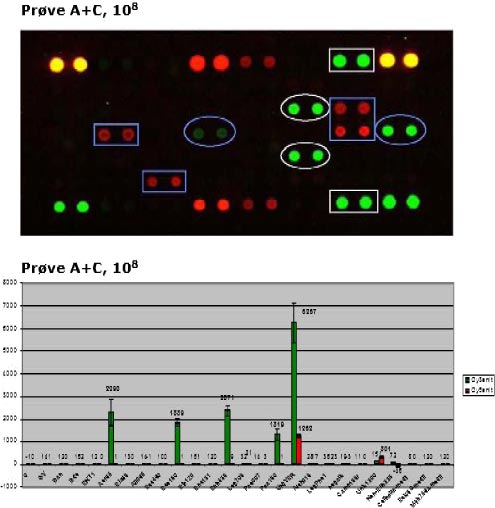
Nederst i figur 5 er illustreret den databehandling, som ligger til grund for alle analyseresultaterne. Ved fluorescens-scanning registreres signalerne fra de Cy3- eller Cy5-molekyler, som PCR produkterne (bundet på chippen) er mærket med. Den efterfølgende databehandling resulterer i en værdi (gennemsnit) for signalintensiteten for hvert enkelt spot på DNA-chippen. Det er disse gennemsnitsværdier, som er afbilledet i figur 5 nederst. Negative kontroller anvendes til at fastlægge en tærskelværdi for hvert slide, og alle signaler over denne tærskelværdi defineres som værende positive, dvs. over baggrundsniveau på det pågældende slide. På figur 5 ses baggrundssignaler som små bitte kolonner lige omkring 0.
Figur 6 viser resultatet af DNA-chip-analyse af suspensionsprøverne A (106) og C (106). Der opnås de forventede (korrekte) signaler for alle patogene bakterier i prøve A (106) (fra venstre): Campylobacter jejuni (hvide kasser), Aeromonas spp. (blå cirkler) og Pseudomonas aeruginosa (blå kasser). Ligeledes viser figur 6 (nederst), at der opnås de forventede signaler for alle patogene bakterier i prøve C (106), fra venstre: Campylobacter jejuni (hvide kasser), Legionella pneumophila /Legionella spp. (grønne kasser) og Salmonella spp. (gule kasser).
Figur 6: Resultat af DNA-chip-analyse af suspensionsprøver: Prøve A (106) (øverst) og prøve C (106) (nederst). Prøve A (106) indeholder Campylobacter jejuni, Aeromonas spp. og Pseudomonas aeruginosa. Prøve C (106) indeholder Campylobacter jejuni, Legionella pneumophila og Salmonella spp.

De øvrige positive signaler på chip-analysen af suspensionsprøve A (106) og C (106) i figur 6 fås fra kontrolproberne UNIVER, Univ1390 og Eub338 (som tidligere beskrevet i afsnit 5.3.2).
Derudover fås falske positive signaler (figur 6, prøve A) fra DNA-proberne Alf968, Arch915 og Non-Eub338 (se figur 1 for placering af prober på chippen). Disse falske positive signaler er velkendte fra tidligere test-forsøg med DNA-chip-prototypen og fremkommer på grund af krydshybridisering til enten den mærkede eller umærkede PCR-produkt-streng. Der ses også falske positive signaler for prøve C (106) i figur 6, (med intensitets-forskelle for specielt Non-Eub338). Derudover ses krydshybridisering til GV-proben (røde spots nederst i højre hjørne og 2. række midt), som beskrevet under kontrolforsøg, afsnit 5.3.1.
I Tabel 4 er summeret alle DNA-chip-analyseresultater af suspensionsprøver og spikede vandprøver. Resultaterne er opdelt i 3 skemaer svarende til analyse af bakterieblanding A (øverst), C (midt) og A+C (nederste skema) indeholdende hhv. 10², 104, 106 og 108 celler pr. patogen.
Tabel 4: Resultaterne af DNA-chip-analyse af i alt 24 suspensionsprøver/ spikede vandprøver. I resultatskemaerne er angivet, hvorvidt en given patogen er påvist, ikke påvist (÷) eller om prøven ikke er analyseret (-).
Prøve A indeholdende Campylobacter, Aeromonas og Pseudomonas
| Antal celler pr. patogen | Campylobacter i prøven | Aeromonas cfu/ml | Pseudomonas cfu/ml | |||
| celle suspension | spiket prøve | celle suspension | spiket prøve | celle suspension | spiket prøve | |
| 10² | ÷ | - | ÷ | - | ÷ | - |
| 104 | påvist | ÷ | påvist | *** | ÷ | ÷ |
| 106 | påvist | påvist | påvist | *** | påvist | påvist |
| 108 | * | * | ** | ** | påvist | påvist |
*) udgik pga. materiale-mangel
**) forurenet med bl.a. Aeromonas (ikke tilsat denne prøve pga. materiale-mangel)
***) forurenet
Prøve C indeholdende Campylobacter, Salmonella og Legionella
| Antal celler pr. patogen | Campylobacter i prøven | Salmonella i prøven | Legionella cfu/ml | |||
| celle suspension | spiket prøve | celle suspension | spiket prøve | celle suspension | spiket prøve | |
| 10² | ÷ | - | ÷ | - | ÷ | - |
| 104 | påvist | ÷ | påvist ** | påvist | ÷ | påvist*** |
| 106 | påvist | påvist | påvist | påvist | påvist | påvist |
| 108 | påvist | * | påvist | påvist | påvist | ÷ |
*) udgik pga. materiale mangel
**) Påvist Salmonella samt forurening med Aeromonas.
***) Legionella påvist med svagt signal fra én probe.
Prøve A+C; Campylobacter, Aeromonas, Pseudomonas, Salmonella og Legionella .
| Campylobact. i prøven | Aeromonas cfu/ml | Pseudomonas cfu/ml | Salmonella i prøven | Legionella cfu/ml | ||||||
| celle susp. | spiket prøve | celle susp. | spiket prøve | celle susp. | spiket prøve | celle susp. | spiket prøve | celle susp. | spiket prøve | |
| 10² | ÷ | - | ÷ | - | ÷ | - | ÷ | - | ÷ | - |
| 104 | ÷** | ÷ | påvist | ÷ | ÷** | ÷ | ÷** | påvist | ÷** | ÷ |
| 106 | påvist | påvist | påvist | påvist | påvist | påvist | påvist | påvist | påvist | ÷* |
| 108 | *** | påvist | påvist | påvist | påvist | påvist | påvist | påvist | påvist | ÷* |
*) ikke påvist, men generelt svagt Cy5- signal på slide, hvilket ses af manglende signal fra Legionella (og Pseudomonas-slægtsprobe).
**) ikke påvist; generelt lave hybridiseringssignaler på dette slide.
***) Campylobacter blev kun tilsat til halvdelen af prøverne pga. materiale-mangel. Campylobacter er påvist i de 108-prøver, hvortil bakterierne er tilsat.
5.3.3 Diskussion og konklusion af DNA-chip-analyseresultater
Resultatet af DNA-chip-analyse af suspensionsprøver og spikede vandprøver indeholdende blandinger af test-organismer viste, at der opnås de forventede (korrekte) signaler for alle patogene bakterier i prøver bestående af 108 og 106 celler pr. patogen pr. prøve.
DNA-chip-metoden kan i dens nuværende design og assay-format ikke påvise specifikke patogener ned til 10² celler pr. prøve, men i nogle tilfælde var det muligt at detektere 104 specifikke patogener, jf. tabel 4.
Til gengæld giver de 3 kontrolprober UNIVER, Eub338 og Univ1390 positive signaler for 104- og 10²-prøverne, dvs. det er muligt at få signaler på DNA-chip-prototypen for disse prøver med kontrolprober, som fanger alle bakterier i prøven. Som eksempel herpå er DNA-chip-analyseresultaterne for kontrolproben Eub338 angivet i tabel 5. Tallene i tabel 5 er Cy3- signalintensiteten (gennemsnit) for kontrolproben Eub338 på DNA-chippen efter analyse af suspensionsprøve A, C og A+C indeholdende 10², 104 og 106 celler pr. patogen pr. prøve. I tabellen er også vist Cy3- signalintensiteten for Camp185r-proben, som er specifik for een patogen (Campylobacter) i dette forsøg.
Negative kontroller anvendes til at fastlægge en tærskelværdi for hver DNA-analyse, og alle signaler over denne tærskelværdi defineres som værende positive, dvs. over baggrundsniveau på den pågældende DNA-chip. Tærskelværdien (Cy3- gennemsnit) for DNA-chips anvendt til analyse af hhv. 10², 104 og 106-prøver er vist i tabel 5, og alle signaler over disse tærskelværdier regnes for positive. Eksempelvis er tærskelværdien (gennemsnit) for alle 10²-prøver i tabel 5 lig med 26, dvs. alle signaler derover er positive. Konklusionen er, at signalerne for Eub338-kontrolproben ligger over eller lige omkring tærskelværdien for 10²-prøverne, dvs. det er muligt at få positive signaler fra disse prøver.
Tabel 5: Resultaterne af DNA-chip-analyse (Cy3- signalværdier) af suspensionsprøver for kontrolproben Eub338 (generel probe) og Camp185r-proben (specifik for Campylobacter).
| Antal celler pr. patogen | Tærskel-værdi (snit) i analysen | Prøve A | Prøve C | Prøve A+C | |||
| Eub338 | Camp185r | Eub338 | Camp185r | Eub338 | Camp185 | ||
| 10² | 26 | 63 | 19 | 16 | 5 | 25 | 13 |
| 104 | 21 | 192 | 38 | 89 | 37 | 49 | 11 |
| 106 | 33 | 6030 | 3864 | 2214 | 2918 | 4173 | 1832 |
Af tabel 5 ses, at det generelt er gældende, at signal-værdien for begge prober er afhængig af antallet af bakterier i prøven, og at signalerne for kontrolproben (som ”fanger” alle bakterier) er højere end signalerne for den specifikke probe Camp185r, (som kun fanger Campylobacter).
5.4 Resultater af prøve A, C og A+C analyseret ved de almindelige dyrkningsmetoder
Resultaterne af de mikrobiologiske analyser af suspensionsprøver og spikede vandprøver af bakterieblanding A, C og A+C (indeholdende hhv. 10², 104, 106 og 108 celler pr. patogen) er summeret i resultatskemaerne i tabel 6 nedenfor. Analyserne for de 5 patogene bakterier er udført ifølge de almindelige dyrkningsmetoder beskrevet i afsnit 2.3.
Tabel 6: Resultater af de mikrobiologiske dyrkningsanalyser af i alt 24 suspensionsprøver/spikede vandprøver. I resultatskemaerne er angivet, hvorvidt en given patogen er påvist, ej påvist (÷) eller om prøven ikke er analyseret (-).
Prøve A indeholdendeCampylobacter, Aeromonas og Pseudomonas
| Antal celler pr. patogen | Campylobacter i prøven | Aeromonas cfu/ml | Pseudomonas cfu/ml | |||
| Celle suspension | spiket prøve | Celle suspension | spiket prøve | celle suspension | spiket prøve | |
| 10² | påvist | påvist | 5,0 × 10¹ | 6 | ÷ | 1,8 × 10¹ |
| 104 | påvist | påvist | 6,7 × 10³ | 1,5 × 104 | 4,0 × 10¹ | 3,7 × 10³ |
| 106 | påvist | påvist | 3,8 × 105 | 8,8 × 105 | 5,0 × 104 | 4,0 × 105 |
| 108 | * | * | * | * | 8,0 × 106 | 9,0 × 107 |
*) udgik pga. materiale-mangel
Prøve C indeholdende Campylobacter, Salmonella og Legionella
| Antal celler pr. patogen | Campylobacter i prøven | Salmonella i prøven | Legionella cfu/ml | |||
| Celle suspension | spiket prøve | Celle suspension | spiket prøve | celle suspension | spiket prøve | |
| 10² | påvist | påvist | påvist | påvist | 1,6 × 10² | 7,8 × 10² |
| 104 | påvist | påvist | ÷ ** | ÷ *** | ingen | 1,9 × 10³ |
| 106 | påvist | påvist | påvist | påvist | 6,0 × 105 | 1,5 × 106 |
| 108 | * | * | påvist | påvist | 2,3 × 107 | 8,0 × 107 |
*) udgik pga. materiale-mangel
**) ingen Salmonella, forurenet med Aeromonas
***) ingen vækst
Prøve A+C indeholdende Campylobacter, Aeromonas, Pseudomonas, Salmonella og Legionella
| Campylobact. i prøven | Aeromonas cfu/ml | Pseudomonas cfu/ml | Salmonella i prøven | Legionella cfu/ml | ||||||
| celle susp. | spiket prøve | celle susp. | spiket prøve | celle susp. | spiket prøve | celle susp. | spiket prøve | celle susp. | spiket prøve | |
| 10² | påvist | påvist | 1,1 × 10² | - | 1 | 3,2 × 10¹ | påvist | påvist | 4,8 × 10¹ | 7,9 × 10² |
| 104 | påvist | påvist | 1,2 × 104 | 1,8 × 104 | 0 | 4,1 × 10³ | påvist | påvist | 9,0 × 10³ | >105 |
| 106 | påvist | påvist | 8,6 × 105 | 1,2 × 106 | 3,5 × 104 | 3,3 × 104* | påvist | påvist | 1,0 × 10² | >106 |
| 108 | påvist | påvist | 7,5 × 106 | 1,3 × 107 | 1,5 × 106 | 6,0 × 107 | påvist | påvist | 5,0 × 107 | 4,9 × 107 |
*) anslået, grundet sammenflydende kolonier er kolonitallet svært at aflæse
5.4.1 Konklusion fra de mikrobiologiske analyseresultater
Konklusionen på de mikrobiologiske analyseresultater er, at dyrkningsmetoderne generelt genfinder de forventede patogene bakterier i blanding A, C og A+C for både suspensionsprøver og spikede vandprøver indeholdende hhv. 108, 106, 104 og 10² celler pr. prøve. Der er enkelte prøver, som ikke har givet de forventede analyseresultater på grund af forskellige tekniske detaljer, jf. kommentarer under resultatskemaerne i tabel 6.
De mikrobiologiske standardmetoder til kvantitativ bestemmelse af hhv. Campylobacter spp. og Salmonella spp. er baseret på en semikvantitativ procedure, som inkluderer fremstilling af 10-folds fortyndinger efterfulgt af en kvalitativ analyse af 10-folds-fortyndingerne. Denne procedure giver en semi-kvantitativ bestemmelse, der ikke direkte kan sammenlignes med de andre kvantitative bestemmelser af Legionella spp., Aeromonas spp. og Pseudomonas spp. i projektet. Desuden kræves større mængder prøvemateriale til semikvantitative analyser. Campylobacter spp. og Salmonella spp. er derfor kun påvist ved en kvalitativ bestemmelse i denne undersøgelse, jf. tabel 5.
De mikrobiologiske analyseresultater og kvantitative bestemmelser af antal bakterier pr. prøve anvendes som udgangspunkt for sammenligning af patogen DNA-chip-prototypen med almindelige dyrkningsmetoder, jf. afsnit 6.
6 Sammenligning af analysemetoder
Analyseresultaterne opnået med DNA-chip-prototypen (tabel 4) skal sammenlignes med resultaterne af de mikrobiologiske dyrkningsmetoder (tabel 6) for både suspensionsprøver og spikede vandprøver for bakterieblanding A, C og A+C.
Generelt er der overensstemmelse imellem resultaterne opnået med DNA-chip-prototypen og de almindelige dyrkningsmetoder for bakterieblandingerne A, C og A+C indeholdende 108 og 106 celler pr. prøve. DNA-chip-metoden kan i dens nuværende design og assay-format ikke detektere specifikke bakterier ned til 10² celler pr. prøve, men i nogle tilfælde var det muligt at detektere 104 specifikke bakterier, jf. tabel 4.
Det skal bemærkes, at prøveforberedelsen (dvs. en ekstra centrifugering af suspensionsprøver og anvendelse af mindre filter-disc fra spikede vandprøver) i begge tilfælde resulterede i en forringet følsomhed for DNA-chip-analysen i dette projekt. Der skal således optimeres på prøveforbehandlingsdelen af projektet for at opnå en forbedret følsomhed.
I figur 8 sammenlignes analyseresultaterne opnået med DNA-chip-prototypen og de mikrobiologiske dyrkningsmetoder for suspensionsprøver og spikede vandprøver indeholdende hhv. 108, 106, 104 og 10² celler pr. patogen pr. prøve. I figuren repræsenterer et grønt felt, at der er 100% (eller meget tæt på 100%) overensstemmelse mellem analysemetoderne. Et gult felt betyder ”delvis overensstemmelse”, mens et rødt felt markerer de prøver, hvor der kun er påvist specifikke bakterier med de mikrobiologiske metoder. Farverne for de enkelte prøver i figur 8 gennemgås i detaljer nedenfor.
Figur 8: Sammenligning af analyseresultaterne opnået med Patogen DNA-chip-prototypen og de mikrobiologiske analysemetoder.

Generelt for prøve A, C og A+C indeholdende 106 eller 102 celler pr. patogen gælder;
- 106: Fuldstændig overensstemmelse mellem DNA-chip-metoden og de mikrobiologiske dyrkningsmetoder (markeret med grønt).
- 10²: Kun påvist specifikke patogene bakterier med de mikrobiologiske dyrkningsmetoder (markeret med rødt). Signaler for positive kontrolprober fås i DNA-chip-analysen, jf. tabel 5.
Kommentarer til prøver indeholdende 108 eller 104 celler pr. patogen;
Prøve A:
- 108: Pseudomonas påvises med begge metoder. Prøven indeholder derudover (ved en fejl) Aeromonas og Salmonella, hvilket kan ses af DNA-chip-analysen.
- 104: I suspensionsprøven påvises Campylobacter og Aeromonas med begge metoder. Pseudomonas påvises kun med dyrkningsmetoden (derfor markeret med gult), hvilket sandsynligvis skyldes, at der kun er tilsat 400 Pseudomonas-bakterier til denne prøve, jf. tabel 6. I den spikede vandprøve detekteres kun specifikke patogener med dyrkningsmetoderne (rød).
Prøve C:
- 108: Legionella (~107 celler) påvises med begge metoder i suspensionsprøven, men kun med dyrkningsmetoden i den spikede vandprøve (derfor markeret med gult i figur 8). Her ses tydeligt effekten af prøveforbehandlingen, som forringer følsomheden for DNA-chip-analysen. Salmonella fundet med begge metoder i begge 108-prøver. Påvisning af Campylobacter i suspensionsprøve er kun udført med DNA-chip-analyse (udgik af de mikrobiologiske analyser).
- 104: Ens resultater med begge metoder for Campylobacter og Legionella. Salmonella og forurening med Aeromonas er fundet med DNA-chip-analysen. Den mikrobiologiske analyse finder også Aeromonas, men ikke Salmonella. I den spikede vandprøve detekteres Legionella med begge metoder, Campylobacter påvises kun med dyrkningsmetoden og Salmonella kun med DNA-chippen.
Prøve A+C:
- 108: Fuldstændig overensstemmelse i mellem de 2 metoder for alle 5 patogener i prøverne med undtagelse af Legionella i den spikede vandprøve, hvilket sandsynligvis skyldes generelt lave signalværdier i denne DNA-chip-analyse, (jf. figur 8**).
- 104: Med dyrkningsmetoderne påvises alle 5 patogener. Med DNA-chip-prototypen detekteres Aeromonas (jf. figur 8*), mens de øvrige signal-værdier for de 5 patogene testorganismer ligger lige omkring detektionsgrænsen, hvilket ses som +/- svage spots på DNA-chippen.
7 Konklusion
I denne undersøgelse er testet en ny hurtiganalyse (DNA-chip-metoden) til påvisning af 5 patogene bakterier (Campylobacter jejuni, Aeromonas hydrophila, Pseudomonas aeruginosa, Legionella pneumophila og Salmonella spp.), som kan være årsag til vandbårne sygdomsudbrud samt forekomme i vandmiljøer som drikkevand og badevand. Analysetiden for DNA-chip-metoden er 1 døgn (inklusiv identifikation af organismerne), hvilket skal sammenlignes med de tilsvarende analyse- og verifikationstider på mellem 2-12 døgn for de mikrobiologiske dyrkningsanalyser.
DNA-chip-prototypen har været igennem mange kontrolforsøg inden opstart af dette projekt. Kontrolforsøgene viste, at enkelte DNA-prober på chippen ikke fungerer optimalt – disse prober er udeladt i dette projekt og vil blive udskiftet ved næste revision af prototypen.
Til gengæld var der ikke udviklet en færdig prøveforberedelsesprocedure til DNA-chip-metoden til analyse af vandprøver inden projektstart, og et sådant udviklingsarbejde lå ikke inden for rammerne af dette projekt. Det blev derfor besluttet at inddrage analyse af suspensionsprøver (=opslemninger af mikroorganismer) i projektet, fordi DNA-chip-analyse af disse prøver ikke ville kræve prøveforberedelse (opkoncentrering). Ved forsøgsstart viste det sig, at start-kulturen af Campylobacter jejuni, Aeromonas hydrophila og Legionella pneumophila ikke var vokset op til den forventede celletæthed, hvilket gav mangel på materiale i forsøget. Som resultat heraf var det ikke muligt at gennemføre DNA-chip-analyse direkte på suspensionsprøver uden prøveforberedelse (se forklaring i afsnit 5.1), og et ekstra opkoncentreringstrin (centrifugering) af suspensionsprøverne blev inkluderet. Dette ekstra opkoncentreringstrin vil betyde et tab af materiale i forsøget (primært for Campylobacter, som er vanskelige at spinde ned), og dermed blev DNA-chip-metodens følsomhed reduceret i forhold til de mikrobiologiske dyrkningsmetoder.
Til prøveforbehandling af spikede vandprøver blev der i projektet valgt en procedure med direkte anvendelse af filtermaterialet i PCR. Fordelen ved denne metode er, at den er hurtig og effektiv over for mange forskellige typer af bakterier. Ulempen er, at proceduren giver mere end en 10x faktors forringelse af DNA-chip-analysens følsomhed ved analyse af de spikede vandprøver, hvilket tydeligt fremgår af resultaterne (generelt lavere signal-værdier for disse analyser sammenlignet med de tilsvarende suspensionsprøver).
Konklusionen på de mikrobiologiske analyseresultater er, at dyrkningsmetoderne generelt genfinder de forventede patogene bakterier i blanding A, C og A+C for både suspensionsprøver og spikede vandprøver indeholdende hhv. 108, 106, 104 og 10² celler pr. prøve. Med DNA-chip-prototypen opnås de forventede (korrekte) signaler for alle patogene bakterier i prøver bestående af 108 og 106 celler pr. patogen pr. prøve.
DNA-chip-metoden kan i dens nuværende design og assay-format ikke detektere specifikke bakterier ned til 10² celler pr. prøve, men i nogle tilfælde var det muligt at detektere 104 specifikke bakterier, jf. tabel 4 (og de ovenfor nævnte forbehold for prøveforbehandling). Signaler for positive kontrolprober fås fra alle prøver (også 10²-prøverne) i DNA-chip-analysen, jf. tabel 5.
Generelt er der overensstemmelse imellem resultaterne opnået med DNA-chip-prototypen og de almindelige dyrkningsmetoder for bakterieblandingerne A, C og A+C indeholdende 108 og 106 celler pr. prøve.
Ud fra resultaterne, opnået i dette projekt, er der behov for følgende:
- DNA-chip-metodens følsomhed skal forbedres, hvis metoden skal anvendes til analyse af patogene bakterier i vand. Optimering af DNA-chip-metodens følsomhed kan gennemføres ved at;
- optimere på selve DNA-chip-assayet (ved at forbedre på mærkningsreaktionen og/eller anvende enkelt-strengede PCR produkter til DNA-chip-analysen) og ved at
- udvikle en prøveforbehandlingsmetode, der er målrettet til DNA-chip-analyse af mange forskellige patogene bakterier i vandprøver.
- Desuden forudsætter anvendelse af DNA-chip-prototypen i praksis, at metoden testes på ”rigtige” vandprøver for at undersøge, hvorvidt forskellige indholdsstoffer (organiske stoffer, metaller etc.) i eksempelvis drikkevand og badevand influerer på analysen.
Bilag A
1 Fastlæggelse af detektionsgrænsen for den PCR-amplifikation af ribosomalt DNA, der indgår i DNA-chip-analysen
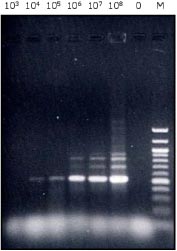
Bilag A:
Fastlæggelse af detektionsgrænsen for 16S PCR amplifiaktionen, der indgår i DNA-chip-analysen. I dette forsøg er der tilsat en fortyndingsrække af rent Salmonella DNA til PCR. Mængden af tilsat DNA er omregnet til antal bakterie-genomer således, at der er tilsat DNA svarende til fra 10³ til 108 celler.
De amplificerede PCR produkter er efterfølgende visualiseret ved agarosegelelektroforese, som illustreret på gelbilledet ovenfor. Resultatet af forsøget viser, at der kan ses PCR produkt ned til 104 celler på agarosegelen, men der er sandsynligvis også PCR produkt i prøven for 10³ celler i mindre mængde, da agarosegelelektroforese ikke er en særlig følsom detektionsmetode.
Version 1.0 December 2006 • © Miljøstyrelsen.