Påvisning af patogene bakterier i vand ved anvendelse af DNA-chip-prototype og mikrobiologiske dyrkningsmetoder
5 Resultater
- 5.1 Fremstilling af suspensionsprøver og prøveforberedelse
- 5.2 Opformering af PCR produkter til DNA-chip-analyse
- 5.3 Resultater opnået med patogen DNA-chip-prototypen
- 5.4 Resultater af prøve A, C og A+C analyseret ved de almindelige dyrkningsmetoder
5.1 Fremstilling af suspensionsprøver og prøveforberedelse
Suspensionskultur-blandinger af A, C og A+C indeholdende hhv. 108, 106, 104 og 10² celler af hver af testorganismerne blev fremstillet, som beskrevet i forsøgsplanen (afsnit 3), bortset fra enkelte afvigelser, som er beskrevet i dette afsnit.
Udgangsmaterialet for forsøget er overnatskulturer af hver af de 5 testorganismer. Ved forsøgsstart viste det sig, at start-kulturen af Campylobacter jejuni, Aeromonas hydrophila og Legionella pneumophila ikke var vokset op til den forventede celletæthed, hvilket gav mangel på materiale i forsøget. Som resultat heraf måtte nogle prøver udgå; således indeholder prøve A (108) kun 108 celler af Pseudomonas aeruginosa, og ikke 108 celler af hhv. Campylobacter jejuni og Aeromonas hydrophila. På tilsvarende vis blev der ikke tilsat Campylobacter jejuni til prøve C (108) og til halvdelen af prøverne A+C (108). Dette gælder for både suspensionsprøver og spikede vandprøver.
Endvidere betød mangel på materiale, at det ikke var muligt at gennemføre DNA-chip-analyse direkte på suspensionsprøver uden prøveforberedelse, som der ellers var lagt op til i den oprindelige forsøgsplan. Suspensionsprøverne skulle efter planen have haft et max. volumen på ca. 60 µl, men dette viste sig ikke at være praktisk muligt, fordi celletætheden af udgangsmaterialet var for lav. Det blev derfor nødvendigt at inkludere et ekstra opkoncentreringstrin (centrifugering) af suspensionsprøverne inden DNA-analyse for at reducere prøvevolumen fra ca. 250 µl til 60 µl. Dette ekstra opkoncentreringstrin vil betyde et tab af materiale (primært for Campylobacter, som er vanskelige at spinde ned), og dermed vil DNA-chip-metodens følsomhed blive reduceret.
Til prøveforbehandling af filtermateriale til DNA-chip-analyse blev der i projektet valgt en procedure med direkte anvendelse af filtermaterialet i PCR. Denne prøveforbehandling kan anvendes på næsten alle bakterier, og prøveforbehandlingen tager kun 10 minutter.
Ulempen ved denne metode er, at prøveforbehandlingen til DNA-chip-analysen på nuværende tidspunkt kun er skaleret til filterdiscs med en diameter på max. 11 mm. Det betyder, at der kun anvendes 7,6 % af det totale filterareal til DNA-chip-analysen, hvilket giver mere end en 10x faktors forringelse af analysens følsomhed. Prøveforbehandlingsteknikken kan opskaleres til at kunne håndtere større filtermaterialer eller der kan anvendes alternative forbehandlingstrin, men et sådant udviklingsarbejde lå ikke inden for rammerne af dette projekt.
5.2 Opformering af PCR produkter til DNA-chip-analyse
Som det fremgår af figur 2 (se afsnit 2.2) foregår en DNA-chip-analyse ved, at ét kort PCR produkt (530 bp) og ét langt PCR produkt (1000 bp) blandes og hybridiseres til DNA-chippen, dvs. der skal fremstilles to PCR produkter fra hver prøve til DNA-chip-analysen.
Alle PCR-produkter visualiseres og kontrolleres ved agarosegelelektroforese inden brug (figur 3). Resultatet af en agarosegelelektroforese illustreret i figur 4 afspejler det generelle resultat for alle PCR-opformeringer af suspensions- og filterdisc-prøver for 530 bp-fragmentet i projektet: Specifikke PCR-produkter af korrekt størrelse blev amplificeret fra A, C og A+C-prøver indeholdende 108 og 106 celler, mens det ikke var muligt at detektere PCR-produkterne fra 104 og 10² celler ved agarosegelelektroforese, dvs. PCR produkterne var til stede i en koncentration, der ligger under detektionsgrænsen på en agarosegel.
Figur 4: Billede af PCR produkter (530 bp fragmenter) efter agarosegel-elektroforese, hvor produkterne kontrolleres inden videre brug til DNA-chip-analyse. (Prøven ”M” længst til højre på gelen er en størrelsesmarkør).
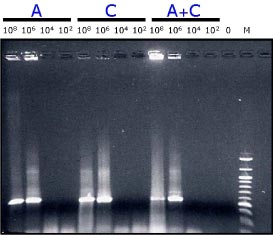
Dette resultat er i overensstemmelse med, at detektionsgrænsen for 16S konsensus PCR-assayet på rent DNA er ca. 10³-104 celler i en agarosegel (se bilag A) samt at prøveforbehandlingen, (dvs. en ekstra centrifugering af suspensionsprøver og anvendelse af mindre filter-disc fra spikede vandprøver) medfører en reduktion af metodens følsomhed.
5.3 Resultater opnået med patogen DNA-chip-prototypen
5.3.1 Kontrolforsøg
Før gennemførelse af DNA-chip-analyse af suspensionsprøver og vandprøver blev DNA-chippen testet i kontrolforsøg: Der blev oprenset rent DNA fra de 5 patogene bakterier i undersøgelsen og test af PCR/DNA-chip-analyse blev udført på kontrolprøver indeholdende kun én patogen samt på prøver indeholdende udvalgte blandinger af de 5 patogene bakterier. Resultaterne viste, at der generelt opnås de forventede signaler med patogen DNA-chip-prototypen.
I enkelte tilfælde kan der forekomme krydshybridisering mellem PCR-produkt fra Legionella og DNA-probe GV for slægten Vibrio. Desuden gav Legionella-DNA-proben generelt svagere signaler på DNA-chip-prototypen end de øvrige prober. Der skal derfor optimeres på Legionella/Vibrio-detektionen før prototypen kan anvendes til specifik detektion af begge disse 2 patogener.
5.3.2 Analyse af prøve A, C og A+C
Figur 5 og 6 viser nogle eksempler på de resultater, der er opnået med patogen DNA-chip-prototypen til analyse af bakterieblanding A, C og A+C.
Som allerede nævnt har patogen DNA-chip-prototypen en standard opsætning med positive kontrol-prober i alle 4 hjørner, der giver positive signaler for hhv. alle bakterier (2 gule spots fra UNIVER-proben i de øverste hjørner) og Eu-bakterier (2 grønne spots fra Eub338-proben i de nederste hjørner), jf. figur 5. De øvrige røde spots på øverste og nederste række på DNA-chippen i figur 5 er signaler fra Univ1390-kontrolproben (til venstre) og et falsk positivt signal fra proben for Archae-bakterier (til højre), som er velkendt fra tidligere testforsøg med denne prototype.
Resultatet af DNA-chip-analyse af suspensionsprøve A+C (108) er illustreret i figur 5, hvor den øverste del af figuren viser et billede af selve DNA-chippen, mens den tilhørende databehandling er illustreret nederst. Af DNA-chippen øverst ses, at der opnås de forventede (korrekte) signaler for alle patogene bakterier i denne prøve A+C (108) (fra venstre på billedet): Legionella pneumophila /Legionella sp. (blå kasser), Aeromonas spp. (blå cirkler), Pseudomonas aeruginosa (hvide cirkler) og Salmonella spp. (hvide kasser).
Figur 5: Resultat af DNA-chip-analyse, hvor patogen DNA-chip-prototypen er anvendt til analyse af suspensionsprøve A+C (108). Prøven indeholder følgende bakterier: Aeromonas spp., Pseudomonas aeruginosa, Salmonella spp. og Legionella pneumophila, serotype 1. Campylobacter jejuni blev ikke tilsat pga. materiale-mangel, jf. afsnit 5.1.
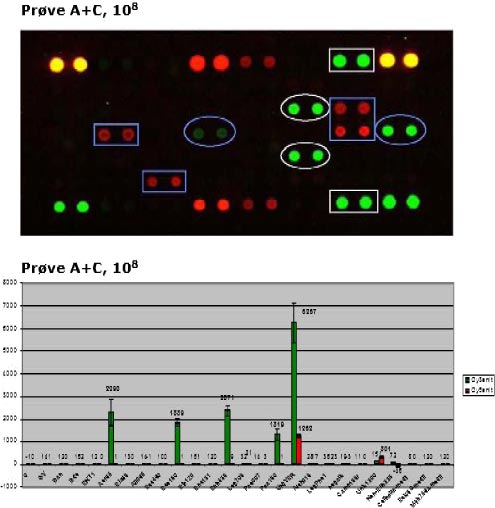
Nederst i figur 5 er illustreret den databehandling, som ligger til grund for alle analyseresultaterne. Ved fluorescens-scanning registreres signalerne fra de Cy3- eller Cy5-molekyler, som PCR produkterne (bundet på chippen) er mærket med. Den efterfølgende databehandling resulterer i en værdi (gennemsnit) for signalintensiteten for hvert enkelt spot på DNA-chippen. Det er disse gennemsnitsværdier, som er afbilledet i figur 5 nederst. Negative kontroller anvendes til at fastlægge en tærskelværdi for hvert slide, og alle signaler over denne tærskelværdi defineres som værende positive, dvs. over baggrundsniveau på det pågældende slide. På figur 5 ses baggrundssignaler som små bitte kolonner lige omkring 0.
Figur 6 viser resultatet af DNA-chip-analyse af suspensionsprøverne A (106) og C (106). Der opnås de forventede (korrekte) signaler for alle patogene bakterier i prøve A (106) (fra venstre): Campylobacter jejuni (hvide kasser), Aeromonas spp. (blå cirkler) og Pseudomonas aeruginosa (blå kasser). Ligeledes viser figur 6 (nederst), at der opnås de forventede signaler for alle patogene bakterier i prøve C (106), fra venstre: Campylobacter jejuni (hvide kasser), Legionella pneumophila /Legionella spp. (grønne kasser) og Salmonella spp. (gule kasser).
Figur 6: Resultat af DNA-chip-analyse af suspensionsprøver: Prøve A (106) (øverst) og prøve C (106) (nederst). Prøve A (106) indeholder Campylobacter jejuni, Aeromonas spp. og Pseudomonas aeruginosa. Prøve C (106) indeholder Campylobacter jejuni, Legionella pneumophila og Salmonella spp.

De øvrige positive signaler på chip-analysen af suspensionsprøve A (106) og C (106) i figur 6 fås fra kontrolproberne UNIVER, Univ1390 og Eub338 (som tidligere beskrevet i afsnit 5.3.2).
Derudover fås falske positive signaler (figur 6, prøve A) fra DNA-proberne Alf968, Arch915 og Non-Eub338 (se figur 1 for placering af prober på chippen). Disse falske positive signaler er velkendte fra tidligere test-forsøg med DNA-chip-prototypen og fremkommer på grund af krydshybridisering til enten den mærkede eller umærkede PCR-produkt-streng. Der ses også falske positive signaler for prøve C (106) i figur 6, (med intensitets-forskelle for specielt Non-Eub338). Derudover ses krydshybridisering til GV-proben (røde spots nederst i højre hjørne og 2. række midt), som beskrevet under kontrolforsøg, afsnit 5.3.1.
I Tabel 4 er summeret alle DNA-chip-analyseresultater af suspensionsprøver og spikede vandprøver. Resultaterne er opdelt i 3 skemaer svarende til analyse af bakterieblanding A (øverst), C (midt) og A+C (nederste skema) indeholdende hhv. 10², 104, 106 og 108 celler pr. patogen.
Tabel 4: Resultaterne af DNA-chip-analyse af i alt 24 suspensionsprøver/ spikede vandprøver. I resultatskemaerne er angivet, hvorvidt en given patogen er påvist, ikke påvist (÷) eller om prøven ikke er analyseret (-).
Prøve A indeholdende Campylobacter, Aeromonas og Pseudomonas
| Antal celler pr. patogen | Campylobacter i prøven | Aeromonas cfu/ml | Pseudomonas cfu/ml | |||
| celle suspension | spiket prøve | celle suspension | spiket prøve | celle suspension | spiket prøve | |
| 10² | ÷ | - | ÷ | - | ÷ | - |
| 104 | påvist | ÷ | påvist | *** | ÷ | ÷ |
| 106 | påvist | påvist | påvist | *** | påvist | påvist |
| 108 | * | * | ** | ** | påvist | påvist |
*) udgik pga. materiale-mangel
**) forurenet med bl.a. Aeromonas (ikke tilsat denne prøve pga. materiale-mangel)
***) forurenet
Prøve C indeholdende Campylobacter, Salmonella og Legionella
| Antal celler pr. patogen | Campylobacter i prøven | Salmonella i prøven | Legionella cfu/ml | |||
| celle suspension | spiket prøve | celle suspension | spiket prøve | celle suspension | spiket prøve | |
| 10² | ÷ | - | ÷ | - | ÷ | - |
| 104 | påvist | ÷ | påvist ** | påvist | ÷ | påvist*** |
| 106 | påvist | påvist | påvist | påvist | påvist | påvist |
| 108 | påvist | * | påvist | påvist | påvist | ÷ |
*) udgik pga. materiale mangel
**) Påvist Salmonella samt forurening med Aeromonas.
***) Legionella påvist med svagt signal fra én probe.
Prøve A+C; Campylobacter, Aeromonas, Pseudomonas, Salmonella og Legionella .
| Campylobact. i prøven | Aeromonas cfu/ml | Pseudomonas cfu/ml | Salmonella i prøven | Legionella cfu/ml | ||||||
| celle susp. | spiket prøve | celle susp. | spiket prøve | celle susp. | spiket prøve | celle susp. | spiket prøve | celle susp. | spiket prøve | |
| 10² | ÷ | - | ÷ | - | ÷ | - | ÷ | - | ÷ | - |
| 104 | ÷** | ÷ | påvist | ÷ | ÷** | ÷ | ÷** | påvist | ÷** | ÷ |
| 106 | påvist | påvist | påvist | påvist | påvist | påvist | påvist | påvist | påvist | ÷* |
| 108 | *** | påvist | påvist | påvist | påvist | påvist | påvist | påvist | påvist | ÷* |
*) ikke påvist, men generelt svagt Cy5- signal på slide, hvilket ses af manglende signal fra Legionella (og Pseudomonas-slægtsprobe).
**) ikke påvist; generelt lave hybridiseringssignaler på dette slide.
***) Campylobacter blev kun tilsat til halvdelen af prøverne pga. materiale-mangel. Campylobacter er påvist i de 108-prøver, hvortil bakterierne er tilsat.
5.3.3 Diskussion og konklusion af DNA-chip-analyseresultater
Resultatet af DNA-chip-analyse af suspensionsprøver og spikede vandprøver indeholdende blandinger af test-organismer viste, at der opnås de forventede (korrekte) signaler for alle patogene bakterier i prøver bestående af 108 og 106 celler pr. patogen pr. prøve.
DNA-chip-metoden kan i dens nuværende design og assay-format ikke påvise specifikke patogener ned til 10² celler pr. prøve, men i nogle tilfælde var det muligt at detektere 104 specifikke patogener, jf. tabel 4.
Til gengæld giver de 3 kontrolprober UNIVER, Eub338 og Univ1390 positive signaler for 104- og 10²-prøverne, dvs. det er muligt at få signaler på DNA-chip-prototypen for disse prøver med kontrolprober, som fanger alle bakterier i prøven. Som eksempel herpå er DNA-chip-analyseresultaterne for kontrolproben Eub338 angivet i tabel 5. Tallene i tabel 5 er Cy3- signalintensiteten (gennemsnit) for kontrolproben Eub338 på DNA-chippen efter analyse af suspensionsprøve A, C og A+C indeholdende 10², 104 og 106 celler pr. patogen pr. prøve. I tabellen er også vist Cy3- signalintensiteten for Camp185r-proben, som er specifik for een patogen (Campylobacter) i dette forsøg.
Negative kontroller anvendes til at fastlægge en tærskelværdi for hver DNA-analyse, og alle signaler over denne tærskelværdi defineres som værende positive, dvs. over baggrundsniveau på den pågældende DNA-chip. Tærskelværdien (Cy3- gennemsnit) for DNA-chips anvendt til analyse af hhv. 10², 104 og 106-prøver er vist i tabel 5, og alle signaler over disse tærskelværdier regnes for positive. Eksempelvis er tærskelværdien (gennemsnit) for alle 10²-prøver i tabel 5 lig med 26, dvs. alle signaler derover er positive. Konklusionen er, at signalerne for Eub338-kontrolproben ligger over eller lige omkring tærskelværdien for 10²-prøverne, dvs. det er muligt at få positive signaler fra disse prøver.
Tabel 5: Resultaterne af DNA-chip-analyse (Cy3- signalværdier) af suspensionsprøver for kontrolproben Eub338 (generel probe) og Camp185r-proben (specifik for Campylobacter).
| Antal celler pr. patogen | Tærskel-værdi (snit) i analysen | Prøve A | Prøve C | Prøve A+C | |||
| Eub338 | Camp185r | Eub338 | Camp185r | Eub338 | Camp185 | ||
| 10² | 26 | 63 | 19 | 16 | 5 | 25 | 13 |
| 104 | 21 | 192 | 38 | 89 | 37 | 49 | 11 |
| 106 | 33 | 6030 | 3864 | 2214 | 2918 | 4173 | 1832 |
Af tabel 5 ses, at det generelt er gældende, at signal-værdien for begge prober er afhængig af antallet af bakterier i prøven, og at signalerne for kontrolproben (som ”fanger” alle bakterier) er højere end signalerne for den specifikke probe Camp185r, (som kun fanger Campylobacter).
5.4 Resultater af prøve A, C og A+C analyseret ved de almindelige dyrkningsmetoder
Resultaterne af de mikrobiologiske analyser af suspensionsprøver og spikede vandprøver af bakterieblanding A, C og A+C (indeholdende hhv. 10², 104, 106 og 108 celler pr. patogen) er summeret i resultatskemaerne i tabel 6 nedenfor. Analyserne for de 5 patogene bakterier er udført ifølge de almindelige dyrkningsmetoder beskrevet i afsnit 2.3.
Tabel 6: Resultater af de mikrobiologiske dyrkningsanalyser af i alt 24 suspensionsprøver/spikede vandprøver. I resultatskemaerne er angivet, hvorvidt en given patogen er påvist, ej påvist (÷) eller om prøven ikke er analyseret (-).
Prøve A indeholdendeCampylobacter, Aeromonas og Pseudomonas
| Antal celler pr. patogen | Campylobacter i prøven | Aeromonas cfu/ml | Pseudomonas cfu/ml | |||
| Celle suspension | spiket prøve | Celle suspension | spiket prøve | celle suspension | spiket prøve | |
| 10² | påvist | påvist | 5,0 × 10¹ | 6 | ÷ | 1,8 × 10¹ |
| 104 | påvist | påvist | 6,7 × 10³ | 1,5 × 104 | 4,0 × 10¹ | 3,7 × 10³ |
| 106 | påvist | påvist | 3,8 × 105 | 8,8 × 105 | 5,0 × 104 | 4,0 × 105 |
| 108 | * | * | * | * | 8,0 × 106 | 9,0 × 107 |
*) udgik pga. materiale-mangel
Prøve C indeholdende Campylobacter, Salmonella og Legionella
| Antal celler pr. patogen | Campylobacter i prøven | Salmonella i prøven | Legionella cfu/ml | |||
| Celle suspension | spiket prøve | Celle suspension | spiket prøve | celle suspension | spiket prøve | |
| 10² | påvist | påvist | påvist | påvist | 1,6 × 10² | 7,8 × 10² |
| 104 | påvist | påvist | ÷ ** | ÷ *** | ingen | 1,9 × 10³ |
| 106 | påvist | påvist | påvist | påvist | 6,0 × 105 | 1,5 × 106 |
| 108 | * | * | påvist | påvist | 2,3 × 107 | 8,0 × 107 |
*) udgik pga. materiale-mangel
**) ingen Salmonella, forurenet med Aeromonas
***) ingen vækst
Prøve A+C indeholdende Campylobacter, Aeromonas, Pseudomonas, Salmonella og Legionella
| Campylobact. i prøven | Aeromonas cfu/ml | Pseudomonas cfu/ml | Salmonella i prøven | Legionella cfu/ml | ||||||
| celle susp. | spiket prøve | celle susp. | spiket prøve | celle susp. | spiket prøve | celle susp. | spiket prøve | celle susp. | spiket prøve | |
| 10² | påvist | påvist | 1,1 × 10² | - | 1 | 3,2 × 10¹ | påvist | påvist | 4,8 × 10¹ | 7,9 × 10² |
| 104 | påvist | påvist | 1,2 × 104 | 1,8 × 104 | 0 | 4,1 × 10³ | påvist | påvist | 9,0 × 10³ | >105 |
| 106 | påvist | påvist | 8,6 × 105 | 1,2 × 106 | 3,5 × 104 | 3,3 × 104* | påvist | påvist | 1,0 × 10² | >106 |
| 108 | påvist | påvist | 7,5 × 106 | 1,3 × 107 | 1,5 × 106 | 6,0 × 107 | påvist | påvist | 5,0 × 107 | 4,9 × 107 |
*) anslået, grundet sammenflydende kolonier er kolonitallet svært at aflæse
5.4.1 Konklusion fra de mikrobiologiske analyseresultater
Konklusionen på de mikrobiologiske analyseresultater er, at dyrkningsmetoderne generelt genfinder de forventede patogene bakterier i blanding A, C og A+C for både suspensionsprøver og spikede vandprøver indeholdende hhv. 108, 106, 104 og 10² celler pr. prøve. Der er enkelte prøver, som ikke har givet de forventede analyseresultater på grund af forskellige tekniske detaljer, jf. kommentarer under resultatskemaerne i tabel 6.
De mikrobiologiske standardmetoder til kvantitativ bestemmelse af hhv. Campylobacter spp. og Salmonella spp. er baseret på en semikvantitativ procedure, som inkluderer fremstilling af 10-folds fortyndinger efterfulgt af en kvalitativ analyse af 10-folds-fortyndingerne. Denne procedure giver en semi-kvantitativ bestemmelse, der ikke direkte kan sammenlignes med de andre kvantitative bestemmelser af Legionella spp., Aeromonas spp. og Pseudomonas spp. i projektet. Desuden kræves større mængder prøvemateriale til semikvantitative analyser. Campylobacter spp. og Salmonella spp. er derfor kun påvist ved en kvalitativ bestemmelse i denne undersøgelse, jf. tabel 5.
De mikrobiologiske analyseresultater og kvantitative bestemmelser af antal bakterier pr. prøve anvendes som udgangspunkt for sammenligning af patogen DNA-chip-prototypen med almindelige dyrkningsmetoder, jf. afsnit 6.
Version 1.0 December 2006, © Miljøstyrelsen.