Påvisning af patogene bakterier i vand ved anvendelse af DNA-chip-prototype og mikrobiologiske dyrkningsmetoder
2 Analysemetoder
- 2.1 Patogen DNA-chip-prototype: Design og opbygning
- 2.2 Beskrivelse af DNA-chip-analysen
- 2.3 Beskrivelse af de anvendte dyrkningsanalyser
- 2.4 Oversigt over analysetider
Denne indledende undersøgelse har fokus på at sammenligne en ny DNA-chip-metode (prototype) med de gældende mikrobiologiske dyrkningsmetoder. I dette afsnit gives en detaljeret beskrivelse af patogen DNA-chip-metoden samt en kort beskrivelse af analyseprincip og analysetider for de mikrobiologiske dyrkningsmetoder, som er anvendt i projektarbejdet.
2.1 Patogen DNA-chip-prototype: Design og opbygning
Bioneer A/S har udviklet en DNA-chip-prototype til simultan detektion af forskellige patogene mikroorganismer. Patogen-DNA-chippen består af et glas-slide, hvorpå der er påsat i alt 24 DNA-prober, som har en længde på mellem 17-23 nukleotider. DNA-proberne er designet udfra bakteriers 16S ribosomale DNA (rDNA), hvilket gør det muligt at opbygge et hierarkisk design af DNA-prober på chippen, som sikrer, at der for hver enkelt bakterie-art detekteres positivt signal fra flere forskellige prober, dvs. en slags intern kvalitetskontrol.
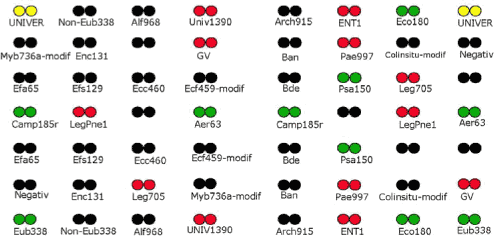
Oversigt over udvalgte bakterier med tilhørende DNA-prober er givet i tabel 1.
Tabel 1: Liste over bakterier med tilhørende 16S rDNA-probe, som er anvendt til udvikling af DNA-chippen (prototype). I kolonnen med 16S rDNA prober er markeret, hvilke prober der aktuelt er anvendt til detektion af de 5 udvalgte patogener i projektet. Hver enkelt DNA-probe på DNA-chippen giver enten Cy3 (grønne signaler) eller Cy5 (røde signaler) alt afhængig af, hvilket af de 2 PCR produkter, som DNA-proben hybridiserer til (se figur 2). De forventede signal-farver for de enkelte DNA-prober er angivet i tabellen med et kryds.

Som det fremgår af tabel 1 veksler probernes specificitetsniveau fra at være specifikke for få arter (f.eks. Psa150 og Camp185r) til at være slægtsspecifikke (f.eks. Aer63 og Leg705) eller generelle (f.eks. UNIVER, Univ1390 og Eub338). Specificitetsniveauet er bestemt ud fra sekvens-analyser med anvendelse af internationale databaser, som indeholder 16S ribosomale DNA-sekvenser fra tusindvis af bakterier.
I projektet anvendtes Camp185r-DNA-proben til påvisning og identifikation af Campylobacter jejuni, men denne DNA-probe kan også detektere C. coli og C. lari, jf tabel 1. Der er 2 prober på DNA-chip-prototypen til detektion af Pseudomonas aeruginosa (Psa150 og Pae997), og ligeledes 2 DNA-prober til detektion af Legionella pneumophila, serotype 1 (LegPne1 og Leg705). Aeromonas hydrophila påvises ved hjælp af Aeromonas-slægtsproben (Aer63). DNA-proben for Enterobacteriaceae (Eco180) anvendes til detektion af Salmonella i dette projekt, fordi DNA-chip-prototypen ikke indeholder en Salmonella-specifik probe.
Tidligere test-forsøg har vist, at Enterobacteriaceae-proben ENT1 ikke virker, hvorfor denne probe ikke inddrages i projektet.
De 3 DNA-prober UNIVER, Univ1390 og Eub338 er anvendt som positive kontroller på DNA-chippen.
I figur 1 er placeringen af de enkelte 16S rDNA-prober på chippen angivet. Patogen DNA-chippen har en standard opbygning med positive kontrol-prober i alle 4 hjørner, der giver positive signaler for hhv. alle bakterier (UNIVER) og Eu-bakterier (Eub338). Derudover er kontrolproben Univ1390 placeret midt på øverste og nederste række, jf. figur 1.
Hver DNA-probe er spottet på DNA-chip-prototypen i duplikat i 2 forskellige positioner, dvs. hver DNA-probe er repræsenteret med 4 spots pr. chip.
Figur 1: Placering af 16S rDNA-prober på patogen DNA-chip (prototype). DNA-prober til detektion af de 5 udvalgte patogene bakterier i projektet samt kontrol-prober er markeret med gul/grøn/rød farve svarende til det forventede signal på DNA-chippen (se tabel 1 og figur 2). Alle øvrige prober er markeret med sort. Spots uden probe-navn er tomme positioner på DNA-chippen.
Fremstillingen af patogen DNA-chips foregik i praksis ved, at DNA-proberne blev spottet på Epoxid-coatede glasslides (Corning) ved hjælp af en Genetix Q-Pix microarrayer.
2.2 Beskrivelse af DNA-chip-analysen
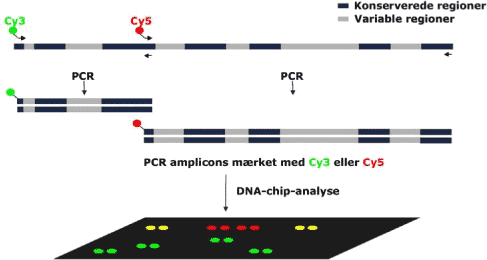
Udgangspunktet for selve DNA-chip-analysen er en enzymatisk polymerase kæde-reaktion (PCR) opformering af DNA vha. en såkaldt konsensus 16S rDNA-PCR reaktion med endemærkede, ribosomale primere.
I praksis gennemføres PCR reaktionen ved amplifikation af hhv. et 530 bp fragment (mærket med Cy3) og et 1000 bp fragment (mærket med Cy5), der tilsammen dækker hele 16S rDNA-regionen (illustreret som den øverste stang i figur 2). De 2 fluorescens-mærkede PCR produkter oprenses, blandes og anvendes til hybridisering til DNA-chippen (figur 2). Resultatet af analysen visualiseres ved fluorescens-scanning, f.eks. på en ArrayWoRx scanner (Applied Precision).
Hvis en given bakterie er til stede i prøven, vil de tilsvarende DNA-prober immobiliseret på DNA-chippen give et positivt Cy3- eller Cy5-signal afhængig af den pågældende DNA-probes placering, dvs. mærkning med to forskellige signal-molekyler giver en intern grøn/rød kvalitetskontrol for diskrimination mellem korrekte og falske-positive signaler i assayet. Gule spots opstår som et mix af grøn og rød, fordi de to PCR produkter hybridiserer lige effektivt til den samme DNA-probe. Dette er tilfældet for en enkelt probe (UNIVER) i det nuværende design af DNA-chippen, fordi proben har et overlap med begge PCR produkter.
Figur 2: Skematisk fremstilling af detektionsprincippet i DNA-chip-analysen.
Patogen-DNA-chippen har været testet under en lang række kontrollerede betingelser, hvor DNA-chippens evne til at give et korrekt respons på blandingsprøver af renkulturer bestående af 2-7 forskellige bakteriearter har været undersøgt og valideret inden starten på dette projekt.
Anvendelse af DNA-chip-metoden til påvisning af i alt 5 patogene bakterier i blanding A, C og A+C (jf afsnit 1.2) er beskrevet i afsnit 3.
2.3 Beskrivelse af de anvendte dyrkningsanalyser
Analyseprincip, arbejdsgangen for at nå frem til det færdige resultat og analysetid er kort beskrevet for hver enkelt mikrobiologisk dyrkningsmetode således, at det er muligt at foretage en samlet vurdering af fordele og ulemper ved DNA-chip-analysen sammenlignet med de mikrobiologiske metoder på baggrund af de metode-tekniske oplysninger i dette afsnit.
2.3.1 Bestemmelse af mesofile Aeromonas arter: NMKL 150:2004
Analyseprincip: Kvantitativ bestemmelse af Aeromonas foretages ved overfladeudsæd af prøven på Stivelse-Ampicillin Agar (SAA) og inkubering ved 37,0°C i 24 timer.
Aflæsning: Aeromonas suspekte kolonier ses som gule eller honningfarvede kolonier med en diameter på 2-3 mm. Kolonierne er omgivet af en lys zone med en bredde på 2-3 mm. Verifikation foretages efter rendyrkning på Luria Bouillon agar(LB agar) uden og med 6% salt. LB-agarpladerne inkuberes ved 37,0°C i 24 timer både aerobt og anaerobt. Før inkubation tilsættes den aerobe skål vibriostatica.
Analysetid: Bestemmelse af Aeromonas-arter efter NMKL 150:2004 tager 2 døgn, inklusiv verifikation af kolonierne, før det endelig resultat foreligger.
2.3.2 Bestemmelse af Pseudomonas aeruginosa: DS 268:1990
Analyseprincip: Kvantitativ bestemmelse af Pseudomonas aeruginosa foretages ved membranfiltrering af prøven, hvorefter filteret anbringes på overfladen af selektivt agar Cetrimid-nalidixinsyre-agar (CN-agar) og inkuberes ved 42°C i 48 timer.
Aflæsning: Fremvoksede kolonier tælles. Pladen anbringes under UV-lys, og der foretages verifikation af fluoriserende kolonier ved subkultivering på mælkeagar. Pladerne inkuberes ved 42°C i 24 timer. Pseudomonas aeruginosa er katalase- og oxidase-positive, nedbryder glucose oxidativt, hydrolyserer kasein (klar zone omkring kolonien på mælkeagar) og danner fluoriserende kolonier.
Analysetid for påvisning og aflæsning af Pseudomonas aeruginosa er 2 døgn plus 1 døgn til subkultivering/verifikation, dvs. i alt 3 døgn, før det endelig resultat foreligger.
2.3.3 Bestemmelse af termofile Campylobacter: ISO 17995:2005
Analyseprincip: Kvalitativ bestemmelse af termofile Campylobacter foretages ved, at prøven membranfiltreres. Filtrene overføres til to selektive opformeringsbouilloner (Bolton bouillon og Preston bouillon), der inkuberes ved 37°C i 2 døgn i mikroaerofil atmosfære. Efter inkubation udstryges fra hver bouillon på overfladen af et selektivt fast substrat, modificeret Charcoal Cephoperazone Desoxycholate Agar, mCCDA, der inkuberes ved 41,5°C i 2 døgn i mikroaerofil atmosfære.
Aflæsning: Suspekte kolonier undersøges for vækst under aerobe forhold. Isolater, der ikke vokser aerobt, undersøges ved mikroskopi. Herefter kan enkeltliggende kolonier verificeres ved subkultivering på blodagarplader med efterfølgende inkubering aerobt og anaerobt ved 41,5°C i 1 døgn. Kolonierne undersøges ved mikroskopi. Campylobacter vokser ikke aerobt, men mikroaerofilt. Campylobacter er stærkt bevægelige, spiralformede (proptrækker-agtige) stave.
Analysetid for termofile Campylobacter inkluderer et selektivt præopformeringstrin på 2 døgn. Den samlede analysetid for termofile Campylobacter er 4 døgn plus ét døgn til verifikation, dvs. i alt 5 døgn, før det endelig resultat foreligger.
2.3.4 Bestemmelse af Salmonella: DS 266:1988
Analyseprincip: Kvalitativ bestemmelse af Salmonella foretages ved, at prøven membranfiltreres. Filtrene overføres til non-selektiv opformeringsbouillon (buffered pepton vand, BPW), der inkuberes ved 37°C i 16-24 timer. Herefter overføres blandingen til 2 selektive opformeringsbouilloner (Rappaport Vassiliadis (RV) bouillon og Müller-Kauffmann bouillon (MK)), der inkuberes ved 42°C i 24 timer. Efter inkubation udstryges fra opformeringen på overfladen af selektive plader, brilliantgrønt-laktose-saccharose-phenolrødt-agar (BLSF) og phenolrødt-mannitol-agar. Pladerne inkuberes ved 37°C i 24 timer.
Aflæsning: På BLSF-agar er typiske kolonier transparente til rødlige, substratet skifter farve fra lys orange til rød. På phenolrødt-mannitol-agar er typiske kolonier gule. Suspekte kolonier rendyrkes på PCA (Plate Count Agar), hvorefter der foretages biokemisk og serologisk verifikation af disse kolonier.
Analysetid: Metoden til påvisning af Salmonella inkluderer to præ-opformeringstrin samt rendyrkning af suspekte kolonier. Den samlede analysetid er 5 døgn, inkl. verifikation, før det endelig resultat foreligger.
2.3.5 Bestemmelse af Legionella: DS 3029:2001
Analyseprincip: En aliquot af prøven (0,1 ml) udsås direkte på GVPC agar (Modificeret Charcoal Yeast Extract agar med selektivt supplement). Den resterende prøve opkoncentreres dels ved membranfiltrering (100x opkoncentrering), dels ved efterfølgende centrifugering (i alt 1000x opkoncentrering) og udsås derefter på GVPC. Pladerne inkuberes ved 37°C. Såfremt pladerne ikke kan aflæses pga. baggrundsvækst udføres syre- og varmebehandling af prøverne før fornyet udsæd.
Aflæsning: GVPC-pladerne aflæses efter ca. 4 og ca. 7 dage. Slutaflæsning foretages efter 10 dage. De fremkomne kolonier tælles og verificeres. Typiske kolonier er konvekse, helrandede, glinsende med kornet overflade. Grå, hvide, blå-violette eller lime-grønne. Nogle kan fluorescere. Kolonierne er små (pin-point) efter 2 – 3 dage, men vokser derefter til ca. 3 – 4 mm. Suspekte kolonier rendyrkes på PCA, hvorefter der foretages biokemisk og serologisk verifikation.
Analysetid for bestemmelse af Legionella er 10 døgn plus 2 døgn til verifikation, før det endelig resultat foreligger.
2.4 Oversigt over analysetider
Overordnet set er der meget stor forskel på den samlede analyse- og verifikationstid for de mikrobiologiske dyrkningsmetoder, jf tabel 2.
Tabel 2: Oversigt over analysetid (døgn) og verifikationstid (døgn) for de anvendte mikrobiologiske dyrkningsmetoder.
| Analysetid, døgn | Verifikation, døgn | |
| Aeromonas spp. | 1 | 1 |
| Pseudomonas aeruginosa | 2 | 1 |
| Campylobacter spp. | 4 | 1 |
| Salmonella spp. | 3 | 2 |
| Legionella spp. | 10 | 2 |
Til sammenligning kan DNA-chip-metoden anvendes til analyse og verifikation af alle 5 udvalgte patogene bakterier inden for 1 døgn.
Version 1.0 December 2006, © Miljøstyrelsen.